 |
Personnes impliquées (A2SI): G. Bertrand, M. Couprie, C. Doublier (ESIEE I5, majeure Informatique).
Partenaires (Hôpital Henri Mondor, Service de Cardiologie): Jean-Luc Dubois- Randé, Thomas Houasse, Jerôme Garot, Olivier Pascal, Emmanuel Teiger, Philippe Garot, Gabriel Pelle.
L'équipe de cardiologie de l'Hôpital Henri Mondor de Créteil dirigée par le professeur Dubois-Randé, et nous-mêmes, avons constitué un groupe imagerie cardiovasculaire. Le projet décrit ci-dessous est le premier projet initialisé par ce groupe en janvier 2002.
Pour l'équipe A2SI, ce projet est dans la continuité de travaux antérieurs en imagerie cardiaque, travaux effectués en partenariat avec l'équipe de Jean-Yves Boire (ERIM, Université d'Auvergne) et la société Sopha-Médical.
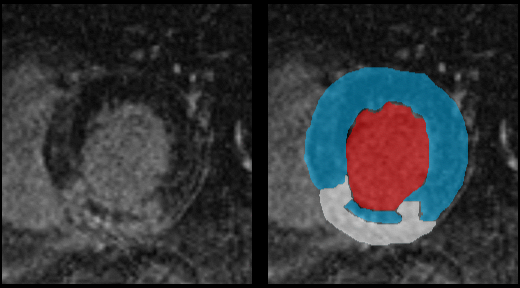
Après un infarctus du myocarde, une partie plus ou moins importante du muscle cardiaque peut se trouver nécrosée. L'imagerie par résonnance magnétique permet de visualiser les parties nécrosées du ventricule gauche grâce à une coloration obtenue par injection d'un produit marqueur dans le sang du patient. Dans la figure 26, on peut voir dans différentes coupes du ventricule gauche le muscle sain (bleu), le volume sanguin à l'intérieur du ventricule (rouge) et les parties nécrosées du muscle (blanc).
Le but de ce projet est de proposer aux chirurgiens un ensemble d'outils informatiques leur permettant d'analyser, de façon plus rapide et plus précise que ne le permettent les techniques actuellement utilisées, les images tri-dimensionnelles fournies par l'IRM. Nous écrivons ci dessous les deux principaux outils en cours de développement.
Segmenteur semi-automatique : cet outil permet au médecin spécialiste de contrôler et de valider une division des différentes coupes composant l'image 3D du myocarde en trois régions : le muscle sain, le muscle nécrosé et le volume sanguin. Un partitionnement préalable est obtenu automatiquement grâce à des opérateurs morphologiques : ligne de partage des eaux et ouvertures par attributs. Les principaux contours des régions d'intérêt sont détectés avec précision et sans intervention manuelle, le spécialiste doit simplement indiquer la nature des différentes zones par quelques clics de souris. En dernier recours, il peut modifier manuellement des contours jugés anormaux. Le programme que nous développons possède une interface utilisateur graphique réalisée avec TCL/TK, ce qui permet une utilisation sous Unix, Linux, Windows.
 |
Générateur de maillage : son rôle est de bâtir un modèle géométrique de la surface des régions segmentées sous la forme d'une triangulation (ensemble de facettes triangulaires liées entre elles par des relations de voisinage). Cet opérateur est basé sur une variante de la technique classique des "marching cubes". Différents algorithmes sont expérimentés permettant de "lisser" la surface des objets géométriques obtenus, pour obtenir d'une part une visualisation plus réaliste, et d'autre part une estimation des volumes que nous espérons plus proches de la réalité (des expériences comprenant des mesures de volumes réels sont prévues pour valider notre méthode).
 |