11. Data frames
La data.frame est la structure de plus haut niveau en R et s’apparente à l’onglet d’un tableur. Les structures de données présentées jusqu’alors (vecteurs, arrays, matrices, listes) sont des structures de plus bas niveau, dont il faut connaître le fonctionnement, mais la plupart du temps, dans une utilisation moderne de R, on travaillera avec des data frames.
Dans le chapitre Un aperçu du fonctionnement de R, on a vu que c’était la structure de données de base utilisé par le package graphique ggplot2. Dans ce chapitre, on va s’intéresser à la façon de construire des data frames et aux différentes fonctions qui permettent de les manipuler.
D’un point de vue fonctionnement interne de R, en fait une data.frame est simplement une list de classe data.frame.
Une data.frame possède les caractéristiques suivantes:
les éléments doivent être des vecteurs (numeric, character, ou logical), des facteurs, des matrices, des listes, ou d’autres data frames.
les matrices, les listes, et les data frames fournissent autant de variables (colonnes) qu’il y a de colonnes dans les matrices, d’éléments dans les listes ou de variables dans les data frames.
les vecteurs numériques ou logique, et les facteurs sont intégrés tels quels. Les vecteurs de chaîne de caractères sont convertis en facteurs.
les vecteurs utilisés pour la construction de la
data.framedoivent avoir la même longueur.les matrices utilisées pour la construction de la
data.framedoivent avoir le même nombre de lignes.les colonnes et les lignes d’une
data.framepeut être extraits suivant la syntaxe utilisée pour les matrices.
11.1. Construire une data frame
Une data.frame peut être construite à partir de vecteurs (satisfaisant les conditions ci dessus) avec la fonction data.frame():
> characters <- c("Walter White", "Jesse Pinkman", "Saul Goodman")
> birthdates <- as.Date(c("1959-09-07","1984-9-14",NA))
> occupations <- c("chemist", "meth manufacturer", "criminal lawyer")
> df <- data.frame(characters, birthdates, occupations)
> df
characters birthdates occupations
1 Walter White 1959-09-07 chemist
2 Jesse Pinkman 1984-09-14 meth manufacturer
3 Saul Goodman <NA> criminal lawyer
Astuce
Une liste dont les éléments satisfont les conventions ci dessus peut être transformée en data.frame avec la fonction as.data.frame().
Dans la plupart des cas, on construira les data frames à partir de données structurées stockées dans un fichier avec la fonction read.table(). On abordera cette façon de faire dans le chapitre Importer des données.
11.2. Utiliser une data.frame
R fournit plusieurs datasets de base permettant de disposer de data frames sans lecture de données externes:
> library(help = "datasets")
Chaque dataset contient des informations descriptives permettant de connaitre la structure des données:
> help(iris)
Le chargement d’un dataset en mémoire est très simple. Utilisons ici le célèbre dataset des iris d’Edgar Anderson:
> data(iris)
> typeof(iris)
[1] "list"
> class(iris)
[1] "data.frame"

Examinons les données brutes dans la console:
> iris
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3.0 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa
5 5.0 3.6 1.4 0.2 setosa
... ... ... ... ... ...
146 6.7 3.0 5.2 2.3 virginica
147 6.3 2.5 5.0 1.9 virginica
148 6.5 3.0 5.2 2.0 virginica
149 6.2 3.4 5.4 2.3 virginica
150 5.9 3.0 5.1 1.8 virginica
On a ainsi un premier aperçu des variables (colonnes) disponibles : la longueur et la largeur des sépales, la longueur et la largeur des pétales, et la variété de la fleur.
11.2.1. Caractéristiques
Les fonctions dim(), colnames() et rownames() permettent de connaitre respectivement la taille, le nom des variables (colonnes) et le nom des observations (lignes). Pour cette dernière caractéristique, le nom est très souvent le numéro de l’observation. On dit aussi l’index:
> dim(iris)
[1] 150 5
> colnames(iris)
[1] "Sepal.Length" "Sepal.Width" "Petal.Length" "Petal.Width" "Species"
> rownames(iris)
[1] "1" "2" "3" "4" "5" "6" ...
On peut extraire les premières lignes:
> head(iris)
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3.0 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa
5 5.0 3.6 1.4 0.2 setosa
6 5.4 3.9 1.7 0.4 setosa
Ou bien les dernières:
> tail(iris)
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
145 6.7 3.3 5.7 2.5 virginica
146 6.7 3.0 5.2 2.3 virginica
147 6.3 2.5 5.0 1.9 virginica
148 6.5 3.0 5.2 2.0 virginica
149 6.2 3.4 5.4 2.3 virginica
150 5.9 3.0 5.1 1.8 virginica
On peut passer un deuxième argument aux fonctions head() et tail() pour indiquer le nombre de ligne souhaitées (6 par défaut). On peut également utiliser un deuxième argument négatif:
> head(iris, -144) # tout sauf les 144 dernières lignes, équivalent à head(iris, 6) ou head(iris)
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3.0 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa
5 5.0 3.6 1.4 0.2 setosa
6 5.4 3.9 1.7 0.4 setosa
> tail(iris, -144)) # tout sauf les 144 premières lignes, équivalent à tail(iris, 6) ou tail(iris)
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
145 6.7 3.3 5.7 2.5 virginica
146 6.7 3.0 5.2 2.3 virginica
147 6.3 2.5 5.0 1.9 virginica
148 6.5 3.0 5.2 2.0 virginica
149 6.2 3.4 5.4 2.3 virginica
150 5.9 3.0 5.1 1.8 virginica
On obtient une vue synthétique de la data.frame avec la fonction str():
> str(iris)
'data.frame': 150 obs. of 5 variables:
$ Sepal.Length: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ...
$ Sepal.Width : num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ...
$ Petal.Length: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ...
$ Petal.Width : num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ...
$ Species : Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 1 1 1 1 1 1 1 ...
et une description statistique avec la fonction summary():
> summary(iris)
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
Min. :4.300 Min. :2.000 Min. :1.000 Min. :0.100 setosa :50
1st Qu.:5.100 1st Qu.:2.800 1st Qu.:1.600 1st Qu.:0.300 versicolor:50
Median :5.800 Median :3.000 Median :4.350 Median :1.300 virginica :50
Mean :5.843 Mean :3.057 Mean :3.758 Mean :1.199
3rd Qu.:6.400 3rd Qu.:3.300 3rd Qu.:5.100 3rd Qu.:1.800
Max. :7.900 Max. :4.400 Max. :6.900 Max. :2.500
Important
Pour les variables (colonnes) numériques, summary() utilise les résultats de la fonction quantile(). Un rafraichissement sur ce que sont des quantiles sur la page Wikipedia.
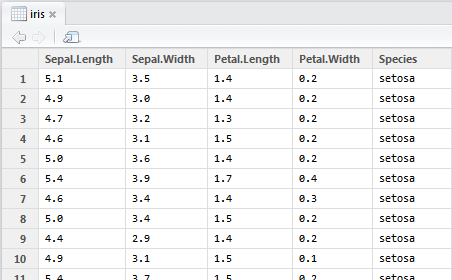
On peut également obtenir une vue plus structurée avec la fonction View() qui met en avance la similitude avec une feuille de tableur:
> View(iris)
11.2.2. Ajouter des données
Une data.frame est une structure de type matricielle et il est possible d’ajouter des lignes et des colonnes avec des fonctions « bas niveau » de type cbind() et rbind(). C’est une bonne pratique de pratiquer une coercition explicite avec la fonction as.data.frame():
> gender <- rep("male",3)
> edf <- cbind(df, as.data.frame(gender))
> edf
characters birthdates occupations gender
1 Walter White 1959-09-07 chemist male
2 Jesse Pinkman 1984-09-14 meth manufacturer male
3 Saul Goodman <NA> criminal lawyer male
L’utilisation de la fonction rbind() nécessite de formatter les lignes à ajouter dans une data.frame avec des noms de variables (colonnes) identiques:
> sw <- list("Skyler White", as.Date(c("1970-08-11")), "car wash manager", "female")
> names(sw) <- names(edf)
> str(sw)
List of 4
$ characters : chr "Skyler White"
$ birthdates : Date[1:1], format: "1970-08-11"
$ occupations: chr "car wash manager"
$ gender : chr "female"
> edf2 <- rbind(edf, as.data.frame(sw))
> edf2
characters birthdates occupations gender
1 Walter White 1959-09-07 chemist male
2 Jesse Pinkman 1984-09-14 meth manufacturer male
3 Saul Goodman <NA> criminal lawyer male
4 Skyler White 1970-08-11 car wash manager female
Evidemment, lorsqu’on ajoute des lignes ou des colonnes à une data.frame, la taille de ce qu’on rajoute doit correspondre à la taille de la data.frame originale:
> nrow(df) == nrow(as.data.frame(gender))
[1] TRUE
> ncol(edf) == ncol(as.data.frame(sw))
[1] TRUE
Avertissement
Cette façon d’ajouter des données à une data.frame est parfaitement valide mais fait appel à des fonctions R core. On utilisera dans le chapitre Transformation de données une méthode plus efficace avec la fonction mutate() du package dplyr de l’éco système Tidyverse.
11.2.3. Sélection de ligne et de colonne
Au delà d’une simple vue globale de la data.frame, on a régulièrement besoin de sélectionner des sous sections particulières pour conduire une analyse poussée sur certaines variables (colonnes) et/ou observations (lignes) qu’il faut sélectionner.
La sélection de colonnes est possible en utilisant l’index de la ou des colonnes concernées:
> iris[,1] # all the rows, column selection by index - output = double
[1] 5.1 4.9 4.7 4.6 5.0 5.4 ...
Puisqu’une data.frame est aussi une list, on peut également utiliser l’opérateur d’indexation de la list:
> iris[[1]] # implicit column selection by index - output = double
> [1] 5.1 4.9 4.7 4.6 5.0 5.4 ...
> class(iris[[1]])
[1] "numeric"
> typeof(iris[[1]])
[1] "double"
Lorsque qu’un seul indice est fourni, il est utilisé pour sélectionner la variable (colonne):
> iris[1] # implicit column selection by index - output = list
Sepal.Length
1 5.1
2 4.9
3 4.7
4 4.6
5 5.0
6 5.4
... ...
> class(iris[1])
[1] "data.frame"
> typeof(iris[1])
[1] "list"
Cette syntaxe est identique à iris[,1]:
> all(iris[1] == iris[,1])
[1] TRUE
On peut également récupérer le contenu de la colonne en indexant avec le nom:
> iris[,"Sepal.Length"] # all the rows, column selection by name - output = double
[1] 5.1 4.9 4.7 4.6 5.0 5.4 ...
> iris["Sepal.Length"] # implicit column selection by name - output = list
Sepal.Length
1 5.1
2 4.9
3 4.7
4 4.6
5 5.0
6 5.4
... ...
> class(iris["Sepal.Length"])
[1] "data.frame"
> typeof(iris["Sepal.Length"])
[1] "list"
On peut également utiliser l’opérateur $ pour simplifier l’écriture:
> iris$Sepal.Length
[1] 5.1 4.9 4.7 4.6 5.0 5.4 ...
La sélection des observations (lignes) obéit au même principe, mais la sélection implicite n’est pas disponible puisqu’elle s’applique aux variables (colonnes):
> iris[1,] # row selection by index, all the columns - output = list
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
> class(iris[1,])
[1] "data.frame"
> typeof(iris[1,])
[1] "list"
Souvent les observations (lignes) ne sont pas nommées ou le sont avec l’index:
> rownames(iris)
[1] "1" "2" "3" "4" "5" "6" "7" "8" "9" "10" "11" "12" "13" "14" "15" "16" "17" "18" "19"
[20] "20" "21" "22" "23" "24" "25" "26" "27" "28" "29" "30" "31" "32" "33" "34" "35" "36" "37" "38"
[39] "39" "40" "41" "42" "43" "44" "45" "46" "47" "48" "49" "50" "51" "52" "53" "54" "55" "56" "57"
[58] "58" "59" "60" "61" "62" "63" "64" "65" "66" "67" "68" "69" "70" "71" "72" "73" "74" "75" "76"
[77] "77" "78" "79" "80" "81" "82" "83" "84" "85" "86" "87" "88" "89" "90" "91" "92" "93" "94" "95"
[96] "96" "97" "98" "99" "100" "101" "102" "103" "104" "105" "106" "107" "108" "109" "110" "111" "112" "113" "114"
[115] "115" "116" "117" "118" "119" "120" "121" "122" "123" "124" "125" "126" "127" "128" "129" "130" "131" "132" "133"
[134] "134" "135" "136" "137" "138" "139" "140" "141" "142" "143" "144" "145" "146" "147" "148" "149" "150"
Et dans ce cas, le nom et l’index étant identiques à un mode près (l’un est numérique, l’autre est une chaîne de caractères), il n’y a aucune différence entre les deux:
> all(iris[1,] == iris["1",])
[1] TRUE
> identical(u1,u2)
[1] TRUE
Bien sûr, on peut mixer la sélection de variables et d’observations:
> iris[1,1]
[1] 5.1
> iris[1,"Sepal.Length"]
[1] 5.1
> iris$Sepal.Length[1]
[1] 5.1
Pour obtenir une plage de valeurs, on passe des vecteurs en lieu et place des simples indices:
> iris[c(1,6),c("Sepal.Length", "Sepal.Width")]
Sepal.Length Sepal.Width
1 5.1 3.5
6 5.4 3.9
> iris[1:6,c("Sepal.Length", "Sepal.Width")]
Sepal.Length Sepal.Width
1 5.1 3.5
2 4.9 3.0
3 4.7 3.2
4 4.6 3.1
5 5.0 3.6
6 5.4 3.9
11.2.4. Indexation logique
On a vu dans le chapitre Vecteurs que les vecteurs logiques sont construit par un prédicat:
> iris$Sepal.Length < 6 & iris$Sepal.Length > 5
[1] TRUE FALSE FALSE FALSE FALSE TRUE ...
On peut utiliser un vecteur logique pour sélectionner les observations satisfaisant à certaines conditions exprimées sur les variables:
> iris[iris$Sepal.Length >= 5.7 & iris$Sepal.Length <= 5.8 & iris$Sepal.Width >=3,]
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
15 5.8 4.0 1.2 0.2 setosa
16 5.7 4.4 1.5 0.4 setosa
19 5.7 3.8 1.7 0.3 setosa
96 5.7 3.0 4.2 1.2 versicolor
L’expression ci dessus contient une data.frame, sur laquelle on peut appliquer l’opérateur de sélection de variable:
> iris[iris$Sepal.Length >= 5.7 & iris$Sepal.Length <= 5.8 & iris$Sepal.Width >=3,]$Petal.Width
[1] 0.2 0.4 0.3 1.2
Pour des questions de lisibilité et de maintenance, c’est une bonne pratique d’écrire la condition de sélection à un seul endroit dans le code et d’y faire référence ensuite:
> fv <- iris$Sepal.Length >= 5.7 & iris$Sepal.Length <= 5.8 & iris$Sepal.Width >=3
> iris[fv,]$Petal.Width
[1] 0.2 0.4 0.3 1.2
11.2.5. Construire un sous ensemble de données
Pour être exhaustif, la fonction subset() (R core) peut être utilisée pour construire un sous ensemble des données de base:
> sdf <- subset(iris, subset=Petal.Length >=1.7, select = c(Sepal.Length, Sepal.Width))
> sdf
Sepal.Length Sepal.Width
6 5.4 3.9
19 5.7 3.8
21 5.4 3.4
24 5.1 3.3
25 4.8 3.4
...
148 6.5 3.0
149 6.2 3.4
150 5.9 3.0
Le paramètre subset est utilisé pour filtrer les observations et le paramètre select pour filtrer les variables.
Avertissement
Les fonctions R core ne sont pas recommandées et on préférera les fonctions dplyr::filter() et dplyr::select() de l’éco système Tidyverse. Elles seront abordées dans le chapitre Transformation de données.
11.2.6. Rechercher les éléments dupliqués et les éléments uniques
Il peut y avoir des données dupliquées dans une data.frame. Selon le contexte de l’étude, on les conservera ou pas, mais il est intéressant de les identifier. La fonction duplicated() retourne un vecteur logique indiquant les éléments dupliqués:
> duplicated(iris)
...
[134] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE TRUE FALSE ...
On se rappelle qu’on peut effectuer un comptage avec la fonction table():
> table(duplicated(iris))
FALSE TRUE
149 1
On affiche l’observation dupliquée en utilisant le vecteur logique retourné par duplicated() comme vecteur d’indexation:
> iris[duplicated(iris),]
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
143 5.8 2.7 5.1 1.9 virginica
Par défaut, duplicated() retourne l’indice de plus haut rang de l’observation dupliquée. On peut cependant lui passer l’argument logique fromLast pour inverser le sens de la recherche et retourner l’élément de plus faible indice:
> iris[duplicated(iris, fromLast = T),]
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
102 5.8 2.7 5.1 1.9 virginica
Récupérer tous les éléments dupliqués est possible en utilisant l’opérateur logique OR appliqué aux deux sens de recherche:
> iris[duplicated(iris) | duplicated(iris, fromLast = T),]
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
102 5.8 2.7 5.1 1.9 virginica
143 5.8 2.7 5.1 1.9 virginica
Avertissement
La fonction R Core duplicated() sera avantageusement remplacé par la fonction dplyr::filter() disponible dans l’éco système Tidyverse. Il faudra utiliser la fonction de comptage (n()>1) comme argument.
Les éléments uniques sont récupérés grâce à la fonction R core unique() qui retourne un sous ensemble de la data.frame originale:
> dim(iris)
[1] 150 5
> u1 <- unique(iris)
> dim(u1)
[1] 149 5
Avertissement
La fonction R Core unique() sera avantageusement remplacé par la fonction dplyr::distinct() disponible dans l’éco système Tidyverse.
11.2.7. Le tri des données
Pour la présentation des données, il est parfois nécessaire de trier une variable de la data.frame selon un ou plusieurs critères. On peut utiliser la fonction R core order() qui retourne, non pas les valeurs elles mêmes, mais un vecteur d’une permutation des indices:
> vpi <- order(iris$Sepal.Length)
> vpi
[1] 14 9 39 43 42 ...
On utilise ce vecteur pour présenter les données triées:
> iris[vpi,]
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
14 4.3 3.0 1.1 0.1 setosa
9 4.4 2.9 1.4 0.2 setosa
39 4.4 3.0 1.3 0.2 setosa
43 4.4 3.2 1.3 0.2 setosa
42 4.5 2.3 1.3 0.3 setosa
...
On observe ci dessus que plusieurs observations ont une variable Sepal.Length identique. On peut passer un deuxième argument à la fonction order(). Il sera utilisé comme critère secondaire pour le tri:
> iris[order(iris$Sepal.Length, iris$Petal.Length),]
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
14 4.3 3.0 1.1 0.1 setosa
39 4.4 3.0 1.3 0.2 setosa
43 4.4 3.2 1.3 0.2 setosa
9 4.4 2.9 1.4 0.2 setosa
42 4.5 2.3 1.3 0.3 setosa
...
La data.frame est ici triée, d’abord par rapport à la variable Sepal.Length, et ensuite les valeurs identiques sont ordonnées avec la variable Petal.Length.
11.3. Des graphiques !
Les structures de données étudiées jusqu’à présent (vecteurs, arrays, matrices) peuvent évidemment être affichés dans des graphiques avec les anciens systèmes graphiques de R base et lattice, mais ils sont limités, complexes à utiliser et incompatibles avec le package graphique qui fait référence : ggplot2.
La structure de données de base pour ggplot2 est la data.frame, dont nous connaissons maintenant les caractéristiques élémentaires.
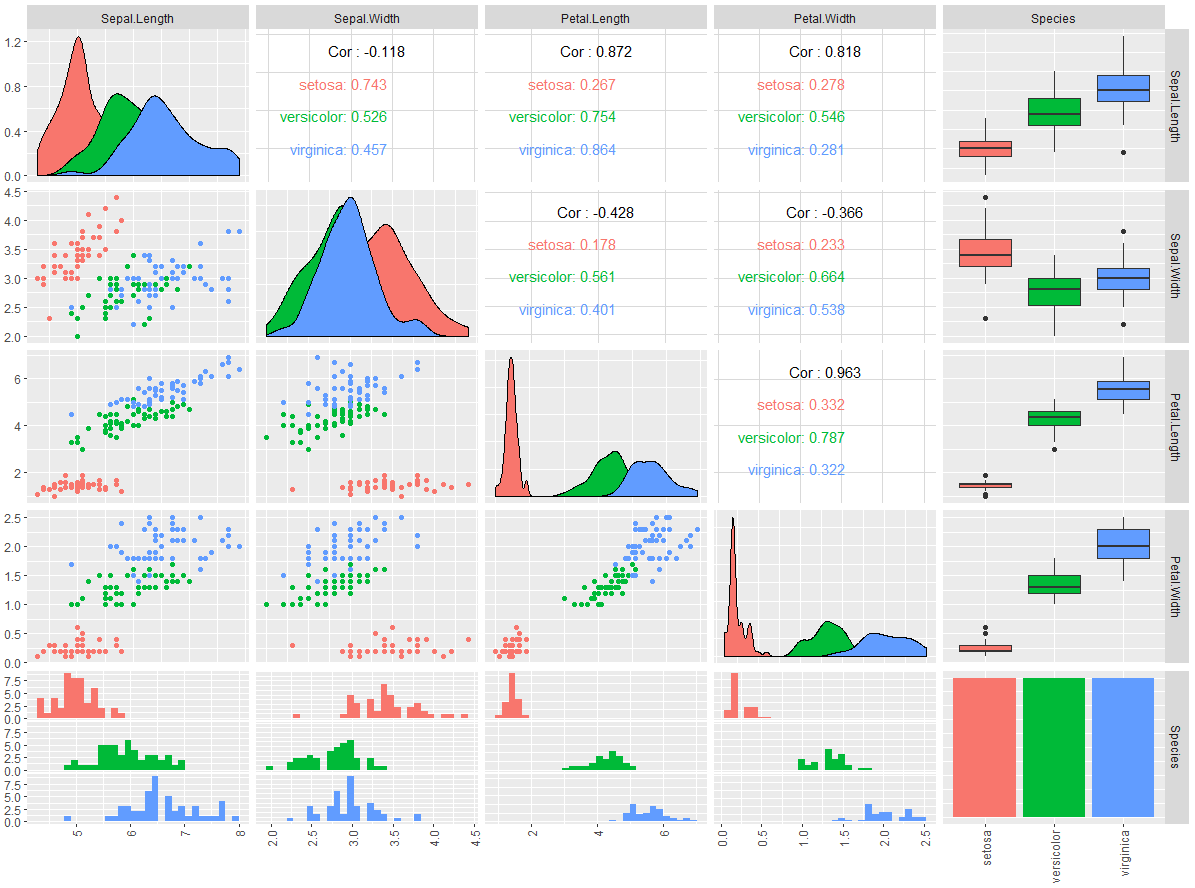
On a déjà eu un aperçu des possibilités étendues de ggplot2 dans le chapitre Un aperçu du fonctionnement de R. Mais ggplot2 peut lui même être étendu avec d’autres packages graphiques, lorsque l’on a à construire des graphiques complexes. On va s’intéresser ici au package GGally et à sa fonction ggpairs() qui permet de produire une série de scatter plots en une seule instruction:
> install.packages("GGally")
...
> library(GGally)
> p <- ggpairs(iris, mapping = aes(color = Species))
> p <- p + theme(axis.text.x=element_text(angle=90,hjust=1,vjust=0.5))
> p
Chaque graphique élémentaire peut évidemment être construit séparément.
11.3.1. Density plot
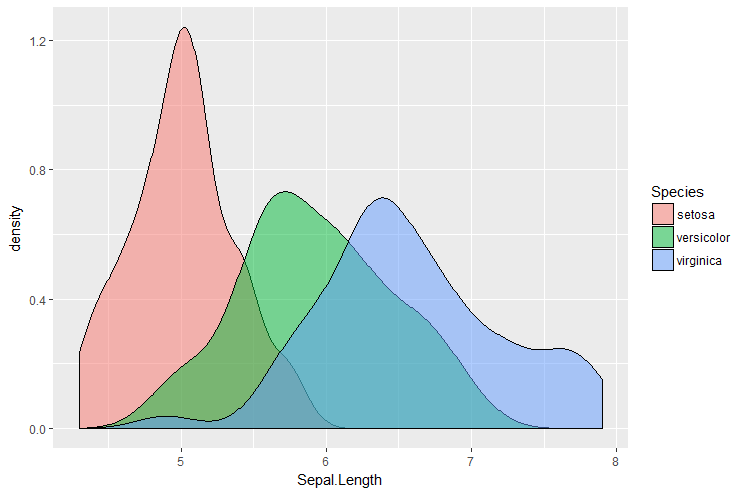
L’affichage de la densité utilise la fonction geom_density() geom. Le paramètre alpha permet de contrôler la transparence:
> ggplot(iris, aes(x=Sepal.Length, fill=Species)) + geom_density(alpha=0.4)
11.3.2. Histogrammes
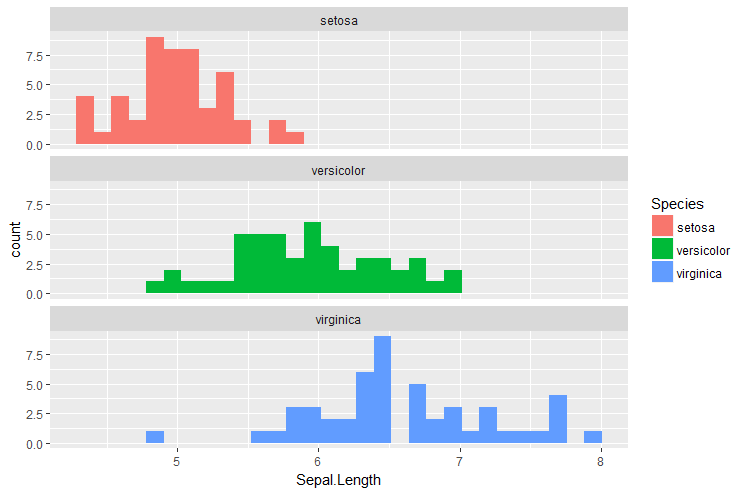
On construit un histogramme avec la fonction geom_histogram() et le layer facetting:
> p <- ggplot(iris, aes(x=Sepal.Length, fill=Species))
> p <- p + geom_histogram()
> p <- p + facet_wrap(~Species, ncol=1)
> p
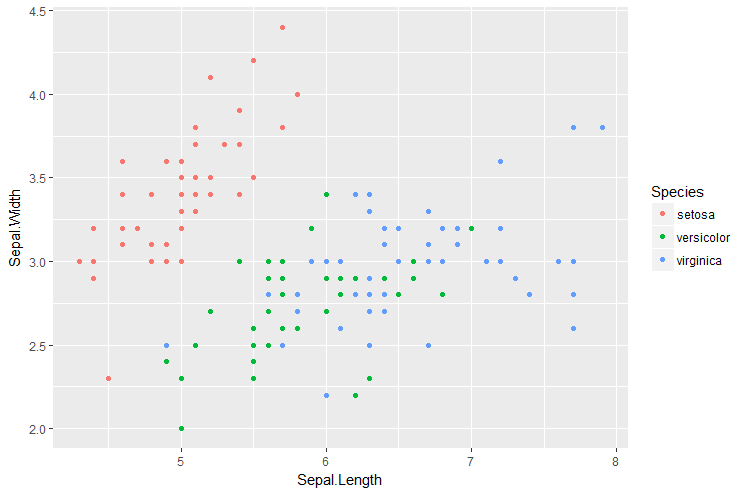
11.3.3. Scatter plots
On a déjà tracé des scatter (ou dot) plots dans le chapitre Un aperçu du fonctionnement de R:
> p <- ggplot(iris, aes(x=Sepal.Length, y=Sepal.Width))
> p <- p + geom_point(aes(color=Species))
> p
11.3.4. Ajouter des labels
Le graphique complexe produit par la fonction GGally::ggpairs() comporte plusieurs types de sous graphiques. Un de ces types est un affichage textuel de données de corrélation.
La corrélation est une mesure de la « similitude » de deux variables (lire cet article pour se rafraichir la mémoire). On calcule une corrélation avec la fonction cor() appliquée à la data.frame entière ou à une sous partie:
> cor(subset(iris, select=-Species)) # toutes les observations, toutes les variables à l'exception de "Species"
Sepal.Length Sepal.Width Petal.Length Petal.Width
Sepal.Length 1.0000000 -0.1175698 0.8717538 0.8179411
Sepal.Width -0.1175698 1.0000000 -0.4284401 -0.3661259
Petal.Length 0.8717538 -0.4284401 1.0000000 0.9628654
Petal.Width 0.8179411 -0.3661259 0.9628654 1.0000000
> cor(subset(iris, Species=="setosa", select=-Species)) # les observations "setosa", toutes les variables à l'exception de "Species"
Sepal.Length Sepal.Width Petal.Length Petal.Width
Sepal.Length 1.0000000 0.7425467 0.2671758 0.2780984
Sepal.Width 0.7425467 1.0000000 0.1777000 0.2327520
Petal.Length 0.2671758 0.1777000 1.0000000 0.3316300
Petal.Width 0.2780984 0.2327520 0.3316300 1.0000000
> cor(subset(iris, Species=="versicolor", select=-Species)) # les observations "versicolor", toutes les variables à l'exception de "Species"
Sepal.Length Sepal.Width Petal.Length Petal.Width
Sepal.Length 1.0000000 0.5259107 0.7540490 0.5464611
Sepal.Width 0.5259107 1.0000000 0.5605221 0.6639987
Petal.Length 0.7540490 0.5605221 1.0000000 0.7866681
Petal.Width 0.5464611 0.6639987 0.7866681 1.0000000
> cor(subset(iris, Species=="virginica", select=-Species)) # les observations "virginica", toutes les variables à l'exception de "Species"
Sepal.Length Sepal.Width Petal.Length Petal.Width
Sepal.Length 1.0000000 0.4572278 0.8642247 0.2811077
Sepal.Width 0.4572278 1.0000000 0.4010446 0.5377280
Petal.Length 0.8642247 0.4010446 1.0000000 0.3221082
Petal.Width 0.2811077 0.5377280 0.3221082 1.0000000
On pourrait aggréger les données de corrélation dans une data.frame avec les fonctions R core (rbind(), cbind() ou merge()) déjà rencontrées mais il est suffisant de connaitre l’existence de ces fonctions un peu obsolètes pour identifier leur rôle dans un code pratique. La bonne pratique sera d’utiliser les fonctions de l’éco système Tidyverse :
tibble::rownames_to_column()dplyr::bind_rows()dplyr::mutate()dplyr::relocate().
Le code Tidyverse pour générer la data.frame est donné ici à titre indicatif (on travaillera spécifiquement sur les fonctions Tidyverse dans le chapitre suivant):
> library(tibble) # un package tidyverse nécessaire
> library(dplyr) # un autre package tidyverse nécessaire
# Construction des data frames de corrélation
> a <- as.data.frame(cor(subset(iris, select=-Species)))
> b <- as.data.frame(cor(subset(iris,Species=="setosa", select=-Species)))
> c <- as.data.frame(cor(subset(iris,Species=="versicolor", select=-Species)))
> d <- as.data.frame(cor(subset(iris,Species=="virginica", select=-Species)))
# Ajout d'une colonne
> a <- rownames_to_column(a,var="SP.WL")
> b <- rownames_to_column(b,var="SP.WL")
> c <- rownames_to_column(c,var="SP.WL")
> d <- rownames_to_column(d,var="SP.WL")
# On "stacke" les data frames élémentaires
> cordf <- bind_rows(a, b, c, d)
# Création du vecteur species
> species <- rep(c("all", "setosa", "versicolor", "virginica"), each=4)
# Ajout d'une variable Species
> cordf <- mutate(cordf, Species=species)
# Déplacement de la variable en première position
> cordf <- relocate(cordf, Species)
La data.frame obtenue:
> cordf
Species SP.WL Sepal.Length Sepal.Width Petal.Length Petal.Width
1 all Sepal.Length 1.0000000 -0.1175698 0.8717538 0.8179411
2 all Sepal.Width -0.1175698 1.0000000 -0.4284401 -0.3661259
3 all Petal.Length 0.8717538 -0.4284401 1.0000000 0.9628654
4 all Petal.Width 0.8179411 -0.3661259 0.9628654 1.0000000
5 setosa Sepal.Length 1.0000000 0.7425467 0.2671758 0.2780984
6 setosa Sepal.Width 0.7425467 1.0000000 0.1777000 0.2327520
7 setosa Petal.Length 0.2671758 0.1777000 1.0000000 0.3316300
8 setosa Petal.Width 0.2780984 0.2327520 0.3316300 1.0000000
9 versicolor Sepal.Length 1.0000000 0.5259107 0.7540490 0.5464611
10 versicolor Sepal.Width 0.5259107 1.0000000 0.5605221 0.6639987
11 versicolor Petal.Length 0.7540490 0.5605221 1.0000000 0.7866681
12 versicolor Petal.Width 0.5464611 0.6639987 0.7866681 1.0000000
13 virginica Sepal.Length 1.0000000 0.4572278 0.8642247 0.2811077
14 virginica Sepal.Width 0.4572278 1.0000000 0.4010446 0.5377280
15 virginica Petal.Length 0.8642247 0.4010446 1.0000000 0.3221082
16 virginica Petal.Width 0.2811077 0.5377280 0.3221082 1.0000000
On peut maintenant extraire les informations de cordf pour les afficher en mode texte:
> p <- ggplot(data=subset(cordf,SP.WL=="Sepal.Width"), aes(x=4,y=c(4:1)))
> p <- p + geom_text(aes(label= sprintf("%18s: %1.3f",Species, Sepal.Length), color=Species), size=8)
> p <- p + theme(legend.position="none")
> p
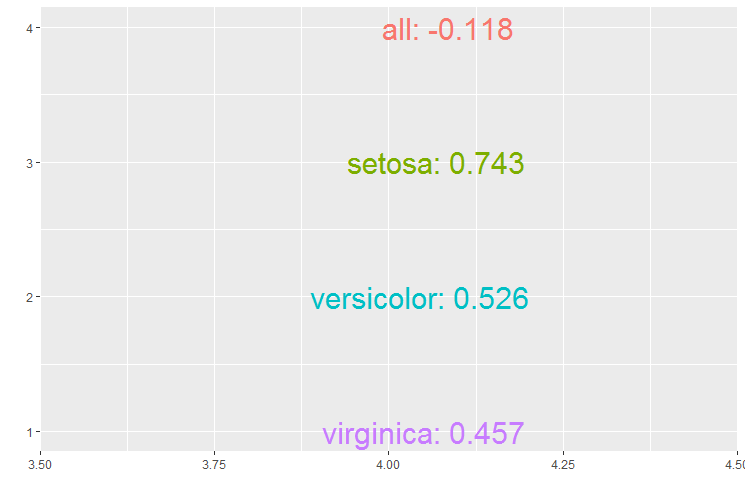
Quelques réglages supplémentaires seraient nécessaires pour formatter le texte, modifier les couleurs, supprimer les labels et la légende, etc.
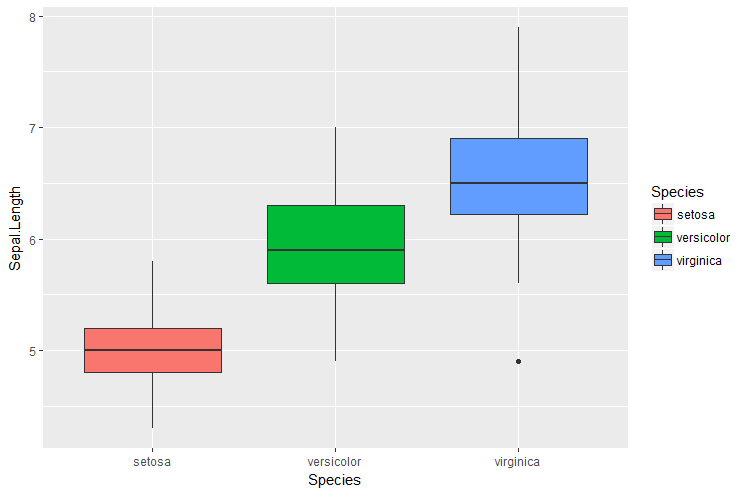
11.3.5. Boxplots
Un boxplot est une façon très compacte de présenter quelques statistiques descriptives (médiane, 1er et 3eme quantiles, valeurs min et max).
> p <- ggplot(iris, aes(x=Species, y=Sepal.Length, fill=Species))
> p <- p + geom_boxplot()
> p
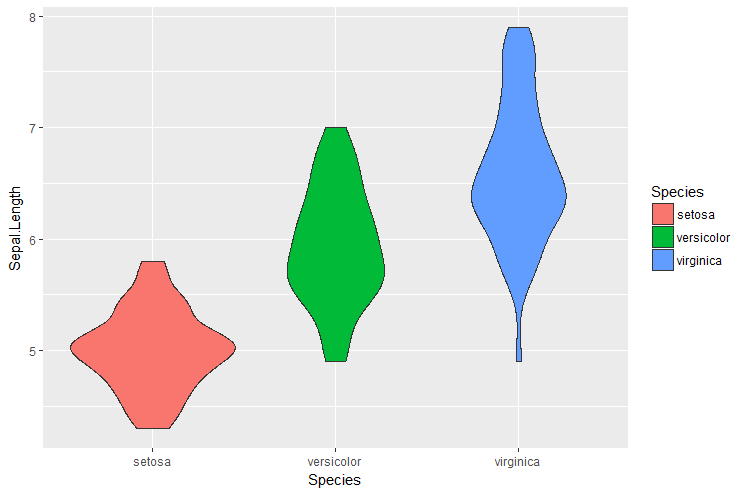
Dans le même esprit, le violin plot fait apparaitre une information sur la forme de la distribution:
> p <- ggplot(iris, aes(x=Species, y=Sepal.Length, fill=Species))
> p <- p + geom_violin()
> p
11.4. Application : la qualité de l’air en Ile de France
L’association AirParif gère des dizaines de capteurs de qualité de l’air en Ile de France. Nous allons nous intéresser ici à celui de Lognes (le plus proche géographiquement d’ESIEE Paris. Les données que l’on va utiliser ont été collectées en avril 2015. Le dataset contient 720 obervations de 5 variables:
Date
Hour
PM10 (µg/m3)
NO2 (µg/m3)
O3 (µg/m3)
Pour pouvoir exploiter pleinement les résultats de l’analyse, on s’intéressera à l’explication fournie ici.
11.4.1. Setup
Démarrer R Studio et fermer tous les projets ouverts avec R Studio > File > Close all
Créer un nouveau projet AirQuality avec R Studio > File > New project... > New Directory > New Project
La data.frame contenant les données est stockée dans l’objet air.RData.
On la charge dans l’environnement avec la commande:
> load("air.RData")
Utiliser les fonctions d’exploration textuelle des données (head(), summary(), str(), etc…) pour une première approche. Identifier les variables présentes, leur nature, les valeurs qu’elles prennent. En un mot, comprendre la totalité des informations présentées:
> summary(air)
date heure PM10.microg.m3. NO2.microg.m3. O3.microg.m3.
Min. :2015-04-01 1 : 30 Min. : 2.00 Min. : 5.0 Min. : 0.00
1st Qu.:2015-04-08 2 : 30 1st Qu.:14.00 1st Qu.: 14.0 1st Qu.: 26.00
Median :2015-04-15 3 : 30 Median :19.00 Median : 22.0 Median : 54.00
Mean :2015-04-15 4 : 30 Mean :21.63 Mean : 30.6 Mean : 51.54
3rd Qu.:2015-04-23 5 : 30 3rd Qu.:27.00 3rd Qu.: 40.5 3rd Qu.: 76.00
Max. :2015-04-30 6 : 30 Max. :65.00 Max. :127.0 Max. :116.00
(Other):540 NA's :10 NA's :1 NA's :3
Utiliser la fonction View() pour une présentation structurée de l’ensemble des données.
La fonction sapply() permet d’appliquer une autre fonction à l’ensemble des variables. Le premier paramètre est la data.frame, le second la fonction à appliquer. Ici la fonction class() retourne le type de chaque colonne:
> sapply(air, class)
date heure PM10.microg.m3. NO2.microg.m3. O3.microg.m3.
"Date" "factor" "numeric" "numeric" "numeric"
Avertissement
La fonction sapply() est une fonction R core. L’éco système Tidyverse fournira des solutions plus efficaces.
La fonction typeof() en donne une représentation interne:
> sapply(air, typeof)
date heure PM10.microg.m3. NO2.microg.m3. O3.microg.m3.
"double" "integer" "double" "double" "double"
On peut observer que R stocke les dates et les heures sous la forme de doubles.
Important
L’analyse des données est un processus impliquant l’inspection, le nettoyage, la transformation et la modélisation de ces données dans le but de découvrir des informations utiles, de suggérer quelques conclusions et d’aider à la prise de décision [Wikipedia]
11.4.2. Inspection
Visualiser les données est généralement un bon point de départ. Un simple plot peut révéler la façon dont les données sont distribuées et donner une bonne idée des premiers éléments statistiques.
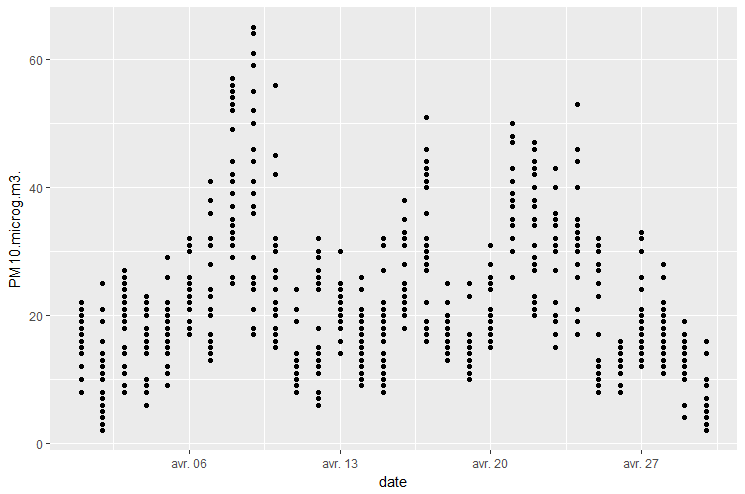
Tracer l’évolution des particules PM10 en fonction de la date. Le graphique obtenu devrait ressembler à ceci.
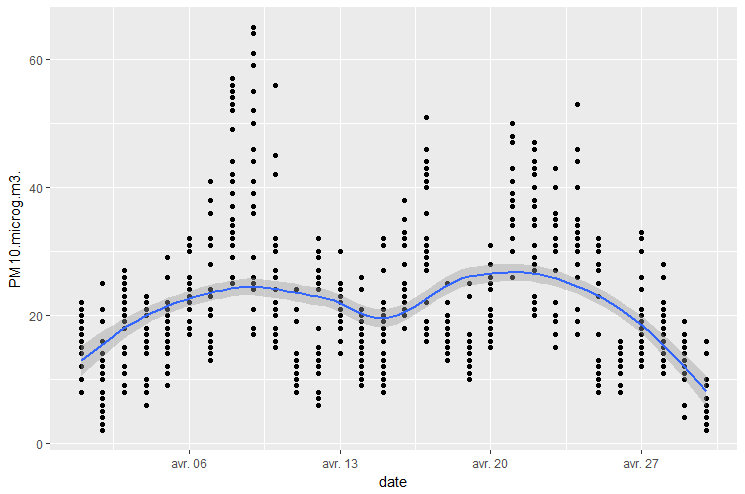
Ajouter une fonction geom_smooth() pour avoir une idée générale de la variation des données.
Il serait intéressant de tracer une série de boxplot comme on l’a fait avec le dataset iris. Cependant la constitution du dataset rend les choses difficiles puisque certaines informations sont stockées dans le nom de la variable. Par exemple la nature du polluant:
> head(air)
date heure PM10.microg.m3. NO2.microg.m3. O3.microg.m3.
1 2015-04-01 1 21 8 79
2 2015-04-01 2 18 9 78
3 2015-04-01 3 16 10 75
4 2015-04-01 4 14 12 69
5 2015-04-01 5 8 21 60
6 2015-04-01 6 10 33 49
A comparer avec le dataset iris pour lequel la variété de fleur est stockée dans une variable:
> head(iris)
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3.0 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa
5 5.0 3.6 1.4 0.2 setosa
6 5.4 3.9 1.7 0.4 setosa
Imaginer la forme que devraient prendre les données de pollution pour se conformer à la structure du dataset iris. En écrire les 5 premières lignes.
11.5. Ressources additionnelles
La documentation de ggplot2 est indispensable pour maitriser la création de graphiques.