12. Transformation de données
Les jeux de données disponibles ne sont pas tous bien formés pour l’analyse. On va s’intéresser dans ce chapitre à la constitution de data frames optimisées pour l’analyse de données et la construction de graphiques avec ggplot2.
12.1. Un dataset mal formé
Comme on l’a vu à la fin du chapitre Data frames, les données de pollution mises à disposition par AirParif ne sont pas structurées de façon optimale et la transformation est un enjeu majeur, sans quoi la présentation et l’analyse en seront au mieux difficiles, au pire impossibles.
Le problème dans ce dataset provient du fait que la nature du polluant dont on mesure la concentration n’est pas accessible comme une observation et ne peut pas être traité automatiquement par les packages de R.
> head(air)
date heure PM10.microg.m3. NO2.microg.m3. O3.microg.m3.
1 2015-04-01 1 21 8 79
2 2015-04-01 2 18 9 78
3 2015-04-01 3 16 10 75
4 2015-04-01 4 14 12 69
5 2015-04-01 5 8 21 60
6 2015-04-01 6 10 33 49
12.2. Un dataset bien formé mais pas optimal
Le dataset iris ne souffre pas du même défaut, la variable catégorielle étant ici une variable à part entière:
> head(iris)
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3.0 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa
5 5.0 3.6 1.4 0.2 setosa
6 5.4 3.9 1.7 0.4 setosa
On parlera ici de dataset tidy puisque toutes les informations sont accessibles à travers des variables. Les règles d’un dataset tidy sont schématisées sur la figure suivante(source: R for Data Science).
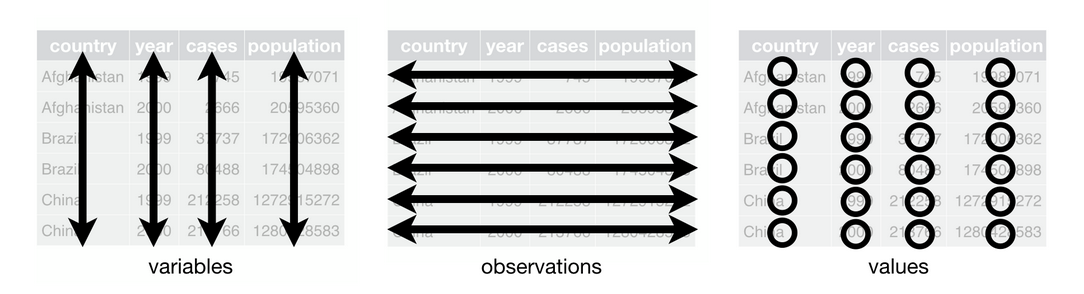
Lire attentivement l’article de Hadley Wickham pour une explication détaillée. Comprendre l’organisation des données est une étape fondamentale pour un traitement performant.
12.3. Un dataset optimal
Les données du dataset iris sont pleinement fonctionnelles (au sens qu’elles respectent l’organisation tidy) parce que l’ensemble des informations sont stockées dans des variables. Ces données sont organisées sous la forme wide, ce qui pourrait s’imager en disant qu’elles sont présentées en mode paysage.
L’autre format possible est le format long (par opposition à wide), et pour poursuivre l’analogie précédente, on pourrait dire que les données sont alors présentées en mode portrait.
Alors que le format wide est intéressant pour l’observation des données par un oeil humain, le format long sera plus adapté à un traitement par la machine et sera privilégié pour plusieurs raisons:
le format
longs’affiche plus facilement dans une fenêtre de taille réduite et évite le scrolling horizontalles données sont structurées en paires clé-valeur, ce qui les rend plus facilement échangeables avec d’autres langages de programmation ou d’autres formats. Pour Python, la structuration clé-valeur est celle des dictionnaires. Pour le web, cette structuration facilite le portage au format JSON, etc…
ggplot2et les packages de Tidyverse sont conçus pour fonctionner avec des data frames utilisant le formatlong.
12.4. La nécessité de modifier le format des données
Il est extrémement rare, voire illusoire, de trouver des données dans le format optimal pour leur traitement. Ca tient à la grande diversité des producteurs de données, ainsi qu’aux différents métiers impliqués.
Les données AirParif utilisées dans le chapitre Data frames en sont une illustration.
Transformer le format des données pourra impliquer une ou plusieurs des actions suivantes:
transformation du format
wideau formatlong(ou réciproquement le cas échéant)sélectionner les observations selon les valeurs d’une ou plusieurs variables
filtrer les variables à retenir pour l’analyse
trier les observations suivant un ou plusieurs critères
créer de nouvelles variables
modifier certaines variables
effectuer une réduction statistique des données
combiner deux ou plusieurs jeux de données
etc…
Certaines de ces opérations ont été effectuées dans les chapitres précédents, souvent avec des opérations à bas niveau ou manipulant les fonctions R core. Nous allons aborder les transformations à plus haut niveau en utilisant les packages Tidyverse.
12.5. Du format wide au format long
Pour le moment la data.frame air est au format wide:
> head(air)
date heure PM10.microg.m3. NO2.microg.m3. O3.microg.m3.
1 2015-04-01 1 21 8 79
2 2015-04-01 2 18 9 78
3 2015-04-01 3 16 10 75
4 2015-04-01 4 14 12 69
5 2015-04-01 5 8 21 60
6 2015-04-01 6 10 33 49
L’objectif est de transformer cette data.frame au format long pour profiter pleinement des possibilités du package ggplot2. La fonction tidyr::pivot_longer() est la fonction appropriée (voir la documentation).
Les principaux paramètres de la fonction tidyr::pivot_longer() sont les suivants:
la
data.frameoriginale au formatwideles variables sélectionnées pour la transformation
names_to: le nom de la nouvelle variable qui remplace les variables sélectionnées pour la transformation. Ces dernières deviennent les valeurs de cette nouvelle variable.values_to: le nom de la nouvelle variable qui va héberger les valeurs des variables transformées
Un exemple pour illustrer ça:
> air_long <- pivot_longer(air, !c("date", "heure"), names_to = "pollutant", values_to = "value")
la
data.frameestairon transforme toutes les variables à l’exception des variables
dateetheurela nouvelle variable (dont les valeurs seront le nom des variables transformées
PM10.microg.m3.,NO2.microg.m3.,O3.microg.m3.) sera nomméepollutantla nouvelle variable qui contiendra les valeurs des variables transformées sera nommée
value
le résultat de la transformation:
> head(air_long)
# A tibble: 6 x 4
date heure pollutant value
<date> <fct> <chr> <dbl>
1 2015-04-01 1 PM10.microg.m3. 21
2 2015-04-01 1 NO2.microg.m3. 8
3 2015-04-01 1 O3.microg.m3. 79
4 2015-04-01 2 PM10.microg.m3. 18
5 2015-04-01 2 NO2.microg.m3. 9
6 2015-04-01 2 O3.microg.m3. 78
> tail(air_long)
# A tibble: 6 x 4
date heure pollutant value
<date> <fct> <chr> <dbl>
1 2015-04-30 23 PM10.microg.m3. 3
2 2015-04-30 23 NO2.microg.m3. 11
3 2015-04-30 23 O3.microg.m3. 61
4 2015-04-30 24 PM10.microg.m3. 2
5 2015-04-30 24 NO2.microg.m3. 8
6 2015-04-30 24 O3.microg.m3. 66
Un aperçu de la data.frame transformée pour visualiser l’ordonnancement de la transformation. Chaque observation de la data.frame initiale est traitée séquentiellement.
> air_long[715:723,]
# A tibble: 9 x 4
date heure pollutant value
<date> <fct> <chr> <dbl>
1 2015-04-10 23 PM10.microg.m3. 21
2 2015-04-10 23 NO2.microg.m3. 12
3 2015-04-10 23 O3.microg.m3. 87
4 2015-04-10 24 PM10.microg.m3. 20
5 2015-04-10 24 NO2.microg.m3. 8
6 2015-04-10 24 O3.microg.m3. 88
7 2015-04-11 1 PM10.microg.m3. 14
8 2015-04-11 1 NO2.microg.m3. 7
9 2015-04-11 1 O3.microg.m3. 85
Cette opération de transformation génère un tibble, une version étendue de la data.frame. Par rapport à la data.frame originale, un tibble apporte des informations supplémentaires, notamment sur le type des variables. On voit ici que le polluant est une simple chaine de caractère alors qu’un facteur serait plus approprié pour effectuer des groupements.
Transformons donc la variable pollutant en facteur:
> air_long$pollutant <- as.factor(air_long$pollutant)
et vérifions la validité de la transformation:
> head(air_long)
# A tibble: 6 x 4
date heure pollutant value
<date> <fct> <fct> <dbl>
1 2015-04-01 1 PM10.microg.m3. 21
2 2015-04-01 1 NO2.microg.m3. 8
3 2015-04-01 1 O3.microg.m3. 79
4 2015-04-01 2 PM10.microg.m3. 18
5 2015-04-01 2 NO2.microg.m3. 9
6 2015-04-01 2 O3.microg.m3. 78
Les valeurs de la variable pollutant ne sont pas très explicites et incluent l’unité de mesure. Remettons un peu d’ordre dans tout ça.
En modifiant l’intitulé des levels du facteur pollutant:
> levels(air_long$pollutant) <- c("NO2","O3", "PM10")
et en transférant l’unité de mesure dans la variable value:
> colnames(air_long)[4] <- "µg.per.m3"
Au final, la data.frame ressemble à:
> head(air_long)
# A tibble: 6 x 4
date heure pollutant µg.per.m3
<date> <fct> <fct> <dbl>
1 2015-04-01 1 PM10 21
2 2015-04-01 1 NO2 8
3 2015-04-01 1 O3 79
4 2015-04-01 2 PM10 18
5 2015-04-01 2 NO2 9
6 2015-04-01 2 O3 78
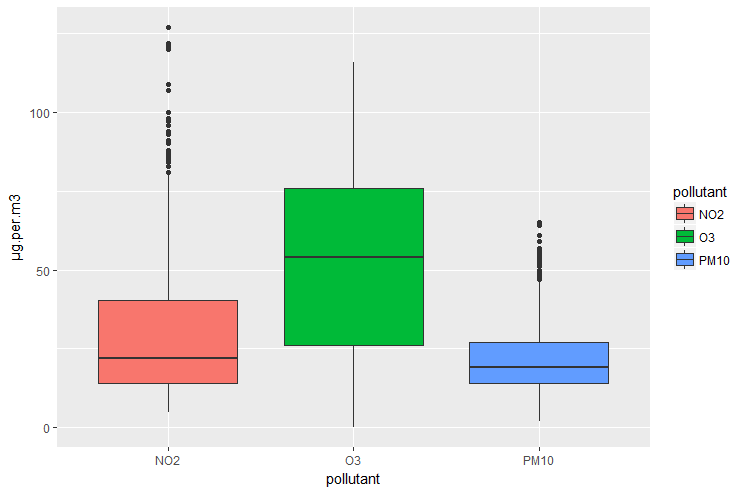
Elle est maintenant prête pour la construction de graphiques:
> p <- ggplot(air_long, aes(x=pollutant, y=µg.per.m3, fill=pollutant))
> p <- p + geom_boxplot()
> p
12.6. Filtrer la data.frame
On peut sélectionner une partie de la data.frame en effectuant une opération de filtrage sur les observations.
Les observations sont filtrées en utilisant un ou plusieurs prédicats impliquant une ou plusieurs variables. Les observations retenues sont celle pour lesquelles l’ensemble des prédicats est évalué à TRUE.
Cette opération est effectuée avec la fonction dplyr::filter(). Voir la documentation du package dplyr.
On peut extraire toutes les mesures d’ozone effectuées à 12:00 pour le mois d’avril 2015:
> filter(air_long, heure==12, pollutant=="O3")
date heure pollutant µg.per.m3
1 2015-04-01 12 O3 77
2 2015-04-02 12 O3 51
3 2015-04-03 12 O3 27
...
28 2015-04-28 12 O3 62
29 2015-04-29 12 O3 76
30 2015-04-30 12 O3 44
On peut construire des prédicats complexes en utilisant les opérateurs logiques:
> filter(air_long, heure==12, pollutant=="O3" | pollutant=="PM10")
date heure pollutant µg.per.m3
1 2015-04-01 12 PM10 20
2 2015-04-02 12 PM10 2
3 2015-04-03 12 PM10 15
...
58 2015-04-28 12 O3 62
59 2015-04-29 12 O3 76
60 2015-04-30 12 O3 44
12.7. Sélection de variables
Le filtrage vu précédemment est l’opération de sélection d’observations (lignes). Les variables (colonnes) sont sélectionnées avec la fonction select():
> select(air_long, -date)
heure pollutant µg.per.m3
1 1 PM10 21
2 2 PM10 18
3 3 PM10 16
4 4 PM10 14
5 5 PM10 8
...
329 17 PM10 9
330 18 PM10 10
331 19 PM10 12
332 20 PM10 17
333 21 PM10 16
[ reached getOption("max.print") -- omitted 1827 rows ]
L’utilisation de l’opérateur de négation vu dans le chapitre Vecteurs est permis.
12.8. Tri des observations
La plupart du temps, les données stockées dans une data.frame sont destinées à être traitées programmatiquement et l’ordonnancement des données n’est pas primordial.
Mais l’examen des données dans la console (avec les fonctions head(), tail(), etc…) peut se trouver facilité par une organisation différente de l’organisation brute du jeu de données.
La fonction arrange() permet ça. Pour le moment, la data.frame air_long est ordonnée par la variable pollutant. S’il est plus judicieux de présenter les données de façon temporelle, on passe les paramètres date et heure (utilisés dans cet ordre) à la fonction arrange():
> arrange(air_long, date, heure)
date heure pollutant µg.per.m3
1 2015-04-01 1 PM10 21
2 2015-04-01 1 NO2 8
3 2015-04-01 1 O3 79
4 2015-04-01 2 PM10 18
5 2015-04-01 2 NO2 9
6 2015-04-01 2 O3 78
...
247 2015-04-04 11 PM10 10
248 2015-04-04 11 NO2 20
249 2015-04-04 11 O3 19
250 2015-04-04 12 PM10 17
[ reached getOption("max.print") -- omitted 1910 rows ]
12.9. Créer ou modifier une variable
La data.frame doit parfois être modifiée pour permettre (ou faciliter) le traitement des données qu’elle contient.
A titre d’exemple la data.frame air_long structure les informations temporelles en deux variables distinctes date et heure. Cette séparation du timestamp en deux variables rend complexe les opérations de calcul temporel. Difficile par exemple de calculer le temps écoulé entre deux observations.
R possède la classe Posixct spécialement conçue pour répondre à ce genre de problème (voir la documentation.). L’objectif est donc de créer un objet de type Posixct à partir des variables date et heure.
La fonction dplyr::mutate() est utilisée pour créer de nouvelles variables ou modifier des variables existantes (voir la documentation).
On modifie la classe de la variable date avec la fonction de coercition as.POSIXct(). Le paramètre format utilise des variables réservées pour définir le formattage à partir de l’objet date. %Y fait référence à l’année au format long, %m fait référence au mois présenté avec un format réduit, etc…:
> air_final <- mutate(air_long, date=as.POSIXct(date, format="%Y%m%d %H%M%S"))
> head(air_final)
# A tibble: 6 x 4
date heure pollutant µg.per.m3
<dttm> <fct> <fct> <dbl>
1 2015-04-01 02:00:00 1 PM10 21
2 2015-04-01 02:00:00 1 NO2 8
3 2015-04-01 02:00:00 1 O3 79
4 2015-04-01 02:00:00 2 PM10 18
5 2015-04-01 02:00:00 2 NO2 9
6 2015-04-01 02:00:00 2 O3 78
On note l’apparition d’un type dttm dans l’affichage du tibble. Ce type est créé par la fonction de coercition as.POSIXct(). La variable date intégre maintenant une information de temps (heure, minute, seconde) mais comme elle n’est pas disponible dans la valeur originale, une valeur par défaut est utilisée:
> sapply(air_final, class)
$date
[1] "POSIXct" "POSIXt"
$heure
[1] "factor"
$pollutant
[1] "factor"
$µg.per.m3
[1] "numeric"
La fonction lubridate::hour() est utilisé pour accéder (en lecture) à l’attribut hour de la variable date.Pour le moment, on ne récupère que les valeurs par défaut mais on a identifié une façon d’accéder à cette information:
> hour(air_final$date)
[1] 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2
...
[ reached getOption("max.print") -- omitted 1160 entries ]
On va utiliser la variable heure à droite de l’opérateur d’affectation pour écrire dans l’attribut hour de la variable date:
> hour(air_final$date) <- as.numeric(air_final$heure)
> head(air_final)
# A tibble: 6 x 4
date heure pollutant µg.per.m3
<dttm> <fct> <fct> <dbl>
1 2015-04-01 01:00:00 1 PM10 21
2 2015-04-01 01:00:00 1 NO2 8
3 2015-04-01 01:00:00 1 O3 79
4 2015-04-01 02:00:00 2 PM10 18
5 2015-04-01 02:00:00 2 NO2 9
6 2015-04-01 02:00:00 2 O3 78
Il reste deux étapes pour obtenir une data.frame parfaitement tidy:
la variable
heureest redondante et doit être suppriméele nom de la variable
datene reflète plus l’information qu’il contient et doit être modifié.
On supprime une variable en lui affectant NULL:
> air_final$heure <- NULL
Les noms de variables sont accessibles avec la fonction colnames():
> colnames(air_final)[1] <- "date_time"
Au final, après ces deux dernières transformations, la data.frame est correctement formée et prête à l’analyse:
> head(air_final)
# A tibble: 6 x 3
date_time pollutant µg.per.m3
<dttm> <fct> <dbl>
1 2015-04-01 01:00:00 PM10 21
2 2015-04-01 01:00:00 NO2 8
3 2015-04-01 01:00:00 O3 79
4 2015-04-01 02:00:00 PM10 18
5 2015-04-01 02:00:00 NO2 9
6 2015-04-01 02:00:00 O3 78
12.10. data.frame ou tibble
On a déjà rencontré l’objet tibble dans le chapitre Un aperçu du fonctionnement de R. En fait les deux structures de données sont très proches l’une de l’autre, et un tibble peut être vu comme une version améliorée de la data.frame, avec essentiellement deux apports/modifications:
l’affichage dans la console est plus efficace et le type des variables est indiqué
la syntaxe de sélection d’une sous partie de la structure est plus stricte. En particulier l’abbréviation du nom des variables ne fonctionne pas.
Passer de l’un à l’autre est direct avec les fonctions de coercition:
> df <- as.data.frame(air_final)
> head(df)
date_time pollutant µg.per.m3
1 2015-04-01 01:00:00 PM10 21
2 2015-04-01 01:00:00 NO2 8
3 2015-04-01 01:00:00 O3 79
4 2015-04-01 02:00:00 PM10 18
5 2015-04-01 02:00:00 NO2 9
6 2015-04-01 02:00:00 O3 78
> class(df)
[1] "data.frame"
La fonction tibble::as_tibble() permet la conversion réciproque:
> library(tibble)
> tb <- as_tibble(df)
> head(tb)
# A tibble: 6 x 3
date_time pollutant µg.per.m3
<dttm> <fct> <dbl>
1 2015-04-01 01:00:00 PM10 21
2 2015-04-01 01:00:00 NO2 8
3 2015-04-01 01:00:00 O3 79
4 2015-04-01 02:00:00 PM10 18
5 2015-04-01 02:00:00 NO2 9
6 2015-04-01 02:00:00 O3 78
La fonction class appliquée à un objet de type tibble montre que c’est une data.frame « augmentée ».
> class(tb)
[1] "tbl_df" "tbl" "data.frame"
En conclusion, travailler avec l’une ou l’autre de ces structures importe peu, mais le tibble est une version plus moderne de la data.frame et ce sera une bonne pratique de l’utiliser.
12.11. Réduction de données
La réduction de données est l’opération qui consiste à représenter un ensemble de valeurs par une seule (moyenne, médiane, écart type, etc…).
Les données, structurées dans un objet tibble, sont maintenant prêtes pour la création de graphique:
> p <- ggplot(air_final, aes(x=date_time, y=µg.per.m3, group=pollutant, color=pollutant))
> p <- p + geom_line()
> p
Ce premier graphique n’est pas très explicite, à cause de la forte densité d’informations et à la périodicité temporelle.
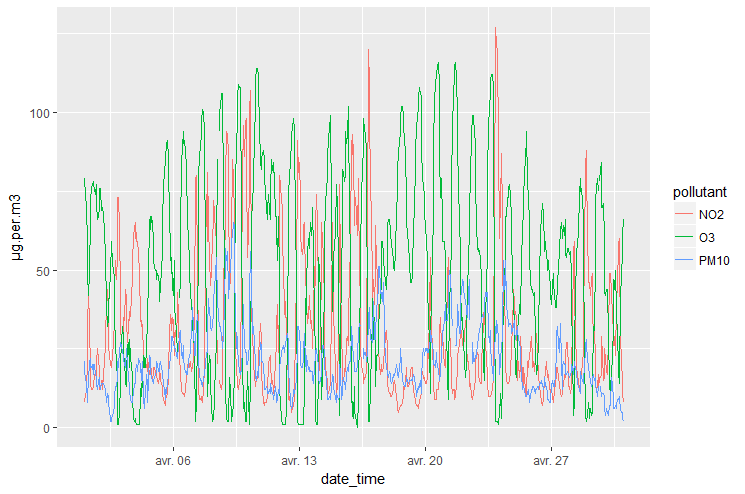
Il nous faut construire des visualisations plus explicites des données de pollution, permettant de tirer des conclusions sans ambigüité. Une première idée est de séparer les jours ouvrés (lundi à vendredi) de ceux du week end. On peut attendre qu’il y ait moins de traffic le week end que la semaine, bien que le samedi puisse être utilisé pour des activités commerciales.
On peut utiliser la fonction lubridate::wday() qui retourne un entier entre 1 et 7 associé au jour de la semaine (dimanche = 1):
> air_WorkingDays <- filter(air_final, wday(date_time)!= 1 & wday(date_time)!= 7)
> air_WeekEnd <- filter(air_final, wday(date_time)== 1 | wday(date_time)== 7)
On peut vérifier la validité de l’opération en récupérant les valeurs uniques des dates référencées dans chacun des sous tibble. Pour celui qui concerne les jours ouvrés, les 4 et 5 avril 2015 sont manquants:
> unique(date(air_WorkingDays$date_time))
[1] "2015-04-01" "2015-04-02" "2015-04-03" "2015-04-06" "2015-04-07"
[6] "2015-04-08" "2015-04-09" "2015-04-10" "2015-04-13" "2015-04-14"
[11] "2015-04-15" "2015-04-16" "2015-04-17" "2015-04-20" "2015-04-21"
[16] "2015-04-22" "2015-04-23" "2015-04-24" "2015-04-27" "2015-04-28"
[21] "2015-04-29" "2015-04-30" "2015-05-01"
Pour celui qui concerne les week ends:
> unique(date(air_WeekEnd$date_time))
[1] "2015-04-04" "2015-04-05" "2015-04-11" "2015-04-12" "2015-04-18"
[6] "2015-04-19" "2015-04-25" "2015-04-26"
Pour calculer la valeur moyenne pour chacun des polluants, on a besoin de deux nouvelles fonctions:
la fonction
dplyr::group_by()utilisé pour effectuer des groupements de donnéesla fonction
dplyr::summarize(), appliquée aux données groupées, pour effectuer la réduction de données proprement dite
La première étape concerne le groupement:
> g <- group_by(air_WorkingDays, pollutant, hour(date_time))
> g
# A tibble: 1,584 x 4
# Groups: pollutant, hour(date_time) [72]
date_time pollutant µg.per.m3 `hour(date_time)`
<dttm> <fct> <dbl> <int>
1 2015-04-01 01:00:00 PM10 21 1
2 2015-04-01 01:00:00 NO2 8 1
3 2015-04-01 01:00:00 O3 79 1
4 2015-04-01 02:00:00 PM10 18 2
5 2015-04-01 02:00:00 NO2 9 2
6 2015-04-01 02:00:00 O3 78 2
7 2015-04-01 03:00:00 PM10 16 3
8 2015-04-01 03:00:00 NO2 10 3
9 2015-04-01 03:00:00 O3 75 3
10 2015-04-01 04:00:00 PM10 14 4
# ... with 1,574 more rows
On note l’information sur le groupement affiché dans l’en tête du tibble.
La seconde étape consiste à effectuer la réduction de données:
> s <- summarize(g, mean=mean(µg.per.m3, na.rm=TRUE))
`summarise()` regrouping output by 'pollutant' (override with `.groups` argument)
> s
# A tibble: 72 x 3
# Groups: pollutant [3]
pollutant `hour(date_time)` mean
<fct> <int> <dbl>
1 NO2 0 45.8
2 NO2 1 37.7
3 NO2 2 33.0
4 NO2 3 32.2
5 NO2 4 36.5
6 NO2 5 49.1
7 NO2 6 54.7
8 NO2 7 52.4
9 NO2 8 39.1
10 NO2 9 33.0
# ... with 62 more rows
Puisque chaque fonction du package dplyr prend une data.frame en entrée et retourne également une data.frame, on peut utiliser l’opérateur de pipe %>% pour écrire de façon compacte et élégante le processus d’analyse de données:
> swd <- air_WorkingDays %>%
+ group_by(pollutant, hour(date_time)) %>%
+ summarize(mean=mean(µg.per.m3, na.rm=TRUE))
`summarise()` regrouping output by 'pollutant' (override with `.groups` argument)
> swd
# A tibble: 72 x 3
# Groups: pollutant [3]
pollutant `hour(date_time)` mean
<fct> <int> <dbl>
1 NO2 0 45.8
2 NO2 1 37.7
3 NO2 2 33.0
4 NO2 3 32.2
5 NO2 4 36.5
6 NO2 5 49.1
7 NO2 6 54.7
8 NO2 7 52.4
9 NO2 8 39.1
10 NO2 9 33.0
# ... with 62 more rows
Important
Lorsqu’on évoque les structures de données produites par les fonctions des packages Tidyverse, on parle de data.frame au sens large. Cette dénomination inclut aussi les tibble.
Cette écriture est particulièrement lisible, car les données produites à gauche du pipe sont utilisées comme entrée pour la fonction située à droite.
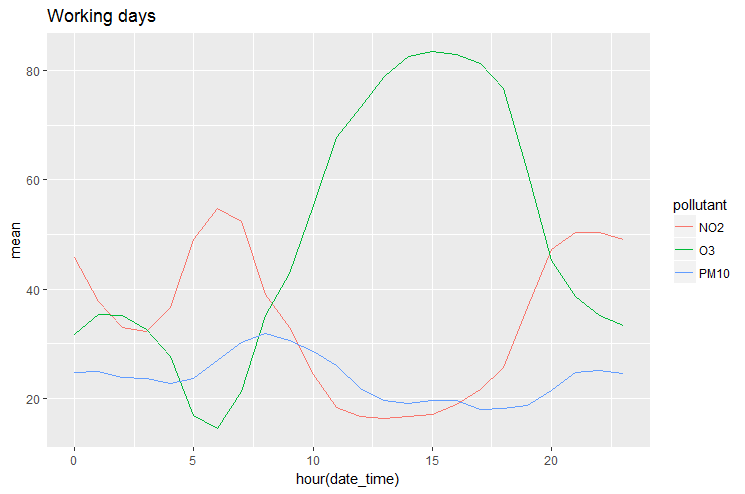
Le graphique est maintenant simple à produire:
> p <- ggplot(swd, aes(x=`hour(date_time)`, y=mean, group=pollutant, color=pollutant))
> p <- p + geom_line()
> p <- p + ggtitle("Working days")
> p
On peut effectuer le même traitement pour les observations effectuées le week end. Il faut d’abord construire la data.frame:
> swe <- air_WeekEnd %>%
+ group_by(pollutant, hour(date_time)) %>%
+ summarize(mean=mean(µg.per.m3, na.rm=TRUE))
`summarise()` regrouping output by 'pollutant' (override with `.groups` argument)
pour afficher les données réduites:
> p <- ggplot(swe, aes(x=`hour(date_time)`, y=mean, group=pollutant, color=pollutant))
> p <- p + geom_line()
> p <- p + ggtitle("Week ends")
> p
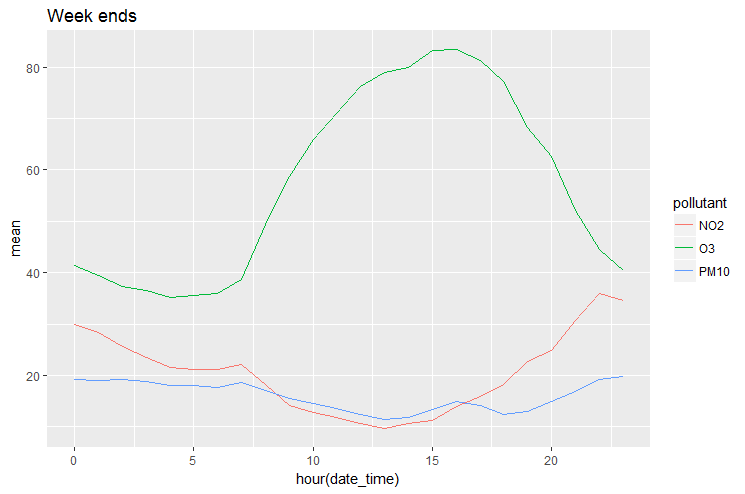
Il est intéressant de superposer les deux graphes. Pour travailler de façon optimale avec ggplot2, la bonne pratique est de construire un seul dataset intégrant une variable logique weekend qui pourrait servir à grouper les données.
12.12. Combiner les data frames
On dispose pour le moment de deux data.frames distinctes contenant pour l’une les données concernant les jours ouvrés, et pour l’autre les données concernant les week end. Cependant l’opération de froupement effectuée par la fonction group_by() a produit une variable dont le nom hour(date_time) n’est pas optimal.
Modifions le avant de combiner les data.frame:
> colnames(swe)[2] <- colnames(swd)[2] <- "hour"
> swe
# A tibble: 72 x 3
# Groups: pollutant [3]
pollutant hour mean
<fct> <int> <dbl>
1 NO2 0 29.9
2 NO2 1 28.4
3 NO2 2 25.6
4 NO2 3 23.5
5 NO2 4 21.6
6 NO2 5 21.1
7 NO2 6 21.1
8 NO2 7 22.1
9 NO2 8 18.2
10 NO2 9 14.2
# ... with 62 more rows
Important
Remarquer ici la double affectation.
On a vu que la fonction dplyr::mutate() pouvait être utilisée pour modifier une variable ou en créer une. On utilise ici cette deuxième possibilité pour ajouter une variable when qui contiendra l’information de la nature du jour de l’observation:
> swd <- mutate(swd, when="WorkingDays")
> swe <- mutate(swe, when="WeekEnd")
Maintenant les data.frame swd et swe sont prêtes à être combinées:
> swe
# A tibble: 72 x 4
# Groups: pollutant [3]
pollutant hour mean when
<fct> <int> <dbl> <chr>
1 NO2 0 29.9 WeekEnd
2 NO2 1 28.4 WeekEnd
3 NO2 2 25.6 WeekEnd
4 NO2 3 23.5 WeekEnd
5 NO2 4 21.6 WeekEnd
6 NO2 5 21.1 WeekEnd
7 NO2 6 21.1 WeekEnd
8 NO2 7 22.1 WeekEnd
9 NO2 8 18.2 WeekEnd
10 NO2 9 14.2 WeekEnd
# ... with 62 more rows
> swd
# A tibble: 72 x 4
# Groups: pollutant [3]
pollutant hour mean when
<fct> <int> <dbl> <chr>
1 NO2 0 45.8 WorkingDays
2 NO2 1 37.7 WorkingDays
3 NO2 2 33.0 WorkingDays
4 NO2 3 32.2 WorkingDays
5 NO2 4 36.5 WorkingDays
6 NO2 5 49.1 WorkingDays
7 NO2 6 54.7 WorkingDays
8 NO2 7 52.4 WorkingDays
9 NO2 8 39.1 WorkingDays
10 NO2 9 33.0 WorkingDays
# ... with 62 more rows
Astuce
Le package dplyr contient beaucoup de fonctions permettant de combiner les data.frame. Pour une vue complète de la combinaison de data.frame, on s’intéressera aux fonctions de la famille xxx_join() décrites dans la documentation de dplyr. Ces fonctions reprennent le vocabulaire des jointures de tables SQL. Une explication assez complète dans l’article Joining Data in R with dplyr.
La fonction dplyr::bind_rows() est ici bien adaptée à notre cas puisque les data.frame sont de structure identique:
> m <- bind_rows(swd, swe)
> m
# A tibble: 144 x 4
# Groups: pollutant [3]
pollutant hour mean when
<fct> <int> <dbl> <chr>
1 NO2 0 45.8 WorkingDays
2 NO2 1 37.7 WorkingDays
3 NO2 2 33.0 WorkingDays
4 NO2 3 32.2 WorkingDays
5 NO2 4 36.5 WorkingDays
6 NO2 5 49.1 WorkingDays
7 NO2 6 54.7 WorkingDays
8 NO2 7 52.4 WorkingDays
9 NO2 8 39.1 WorkingDays
10 NO2 9 33.0 WorkingDays
# ... with 134 more rows
Comme le dit le proverbe, data.frame bien préparée, production de graphiques facilitée ;-)
> p <- ggplot(data=m, aes(x=hour, y=mean, color=pollutant, linetype=when))
> p <- p + geom_line(size=1)
> p <- p + ggtitle("Air pollution in Lognes (A4)")
> p
Le graphique montre très clairement l’évolution de la moyenne de chacun des polluants. Pour étendre l’étude, il serait intéressant d’utiliser les données météorologiques correspondantes, dont on sait qu’elles ont un impact fort.
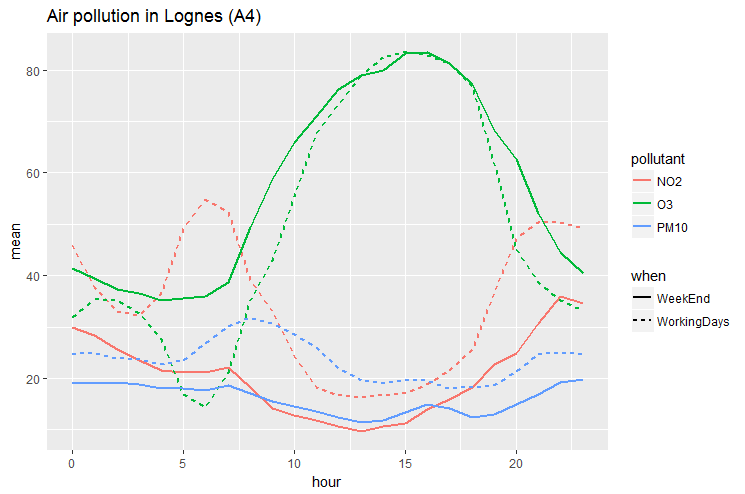
12.13. Application : population urbaine
Reprenons les données des 20 plus grandes villes françaises utilisées dans les chapitres précédents. Ces données ont été structurées dans une matrix dont on a une vue partielle ci dessous:
Angers Bordeaux Brest Dijon Grenoble Le.Havre Le.Mans ...
1962 115273 278403 136104 135694 156707 187845 132181 ...
1968 128557 266662 154023 145357 161616 207150 143246 ...
1975 137591 223131 166826 151705 166037 217882 152285 ...
1982 136038 208159 156060 140942 156637 199388 147697 ...
1990 141404 210336 147956 146703 150758 195854 145502 ...
1999 151279 215363 149634 149867 153317 190905 146105 ...
2007 151108 235178 142722 151543 156793 179751 144164 ...
2012 149017 241287 139676 152071 158346 173142 143599 ...
Pour tracer quelques graphiques, ggplot2 nécessite des objets de type data.frame. Utiliser la fonction as.data.frame() pour transformer cette matrice (construite dans le chapitre Tableaux multidimensionnels) en une data.frame.
Cette data.frame est fonctionnelle au sens où toutes les données sont accessibles mais elle présente plusieurs défauts:
elle est au format
widequi n’est pas optimal pour la construction de graphiquesune donnée fondamentale (l’année) n’est pas une variable mais le nom de l’observation. Elle n’est pour le moment pas utilisable pour effectuer des groupements de données.
Appliquer les transformations nécessaires pour supprimer ces deux inconvénients. On nommera dfl la data.frame transformée.
Après transformation, la data.frame dfl se présente maintenant de la façon suivante:
> head(dfl, 10)
# A tibble: 10 x 3
year city pop
<fct> <fct> <dbl>
1 1962 Angers 115273
2 1962 Bordeaux 278403
3 1962 Brest 136104
4 1962 Dijon 135694
5 1962 Grenoble 156707
6 1962 Le.Havre 187845
7 1962 Le.Mans 132181
8 1962 Lille 239955
9 1962 Lyon 535746
10 1962 Marseille 778071
On prêtera une attention particulière au type de chacune des variables.
Cette data.frame est maintenant parfaitement formattée pour la construction de graphiques.
12.13.1. Un premier graphique
Tracer l’évolution de la population de Bordeaux. Le graphique doit être similaire à celui ci dessous.
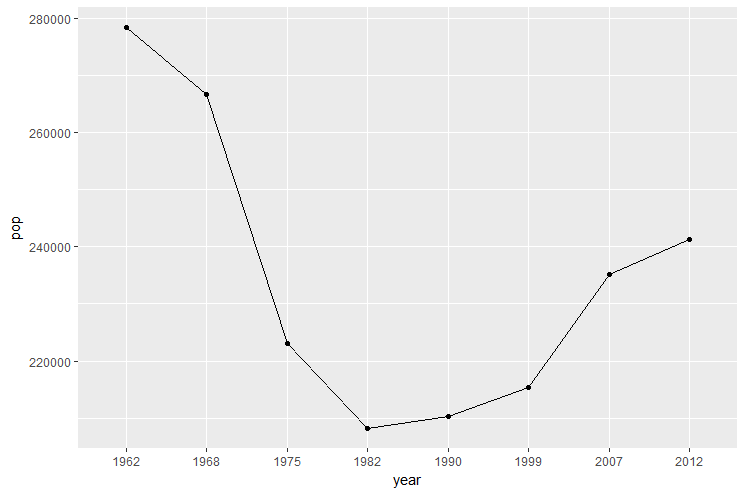
Astuce
Comme year est une variable catégorielle, le paramètre group doit être passé à la fonction aes() pour indiquer à geom_line() que les points doivent être reliés entre eux. Sinon, on obtient le message « Each group consists of only one observation ».
Important
ggplot2 met en oeuvre un mécanisme d’héritage entre les différents layers. Les paramètres définis dans un layer de plus bas niveau sont disponibles dans un layer de plus haut niveau qui hérite des propriétés des layers inférieurs. Un paramètre hérité peut être « écrasé » dans un layer supérieur. Ainsi, le paramètre mapping peut être indifférement passé à la fonction ggplot(), ou à la fonction geom_xxx().
12.13.2. Modifier l’esthétique
On peut modifier la façon dont les données sont affichées en passant des paramètres spécifiques aux fonctions de la famille geom_xxx(). Lire cet article.
Passer les paramètres nécessaires à geom_line() et geom_point() pour obtenir un graphique similaire à celui ci dessous:
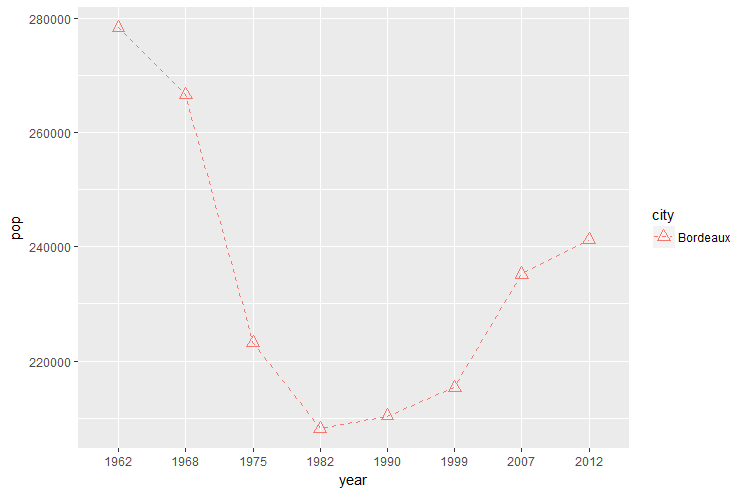
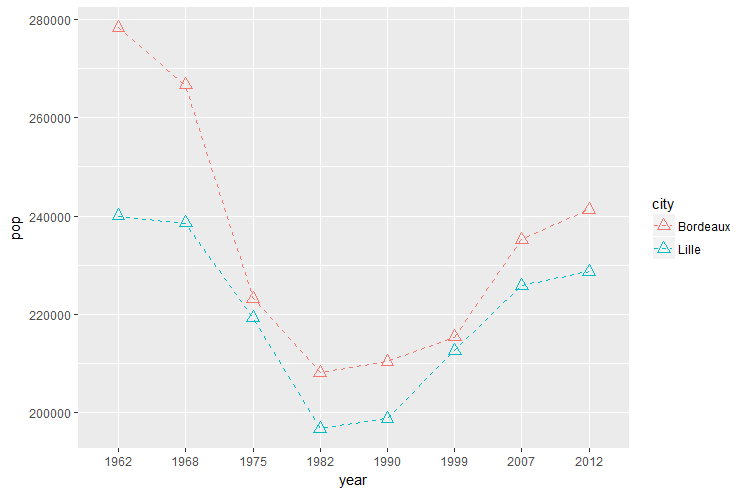
12.13.3. Plusieurs courbes sur le même graphique
La structuration apportée par ggplot2 permet de construire des graphes modulaires.
Construire un graphe similaire à celui ci dessous, en modifiant le filtrage effectué sur la data.frame. Définir le maximum de paramètres dans le layer de plus bas niveau avec la fonction ggplot().
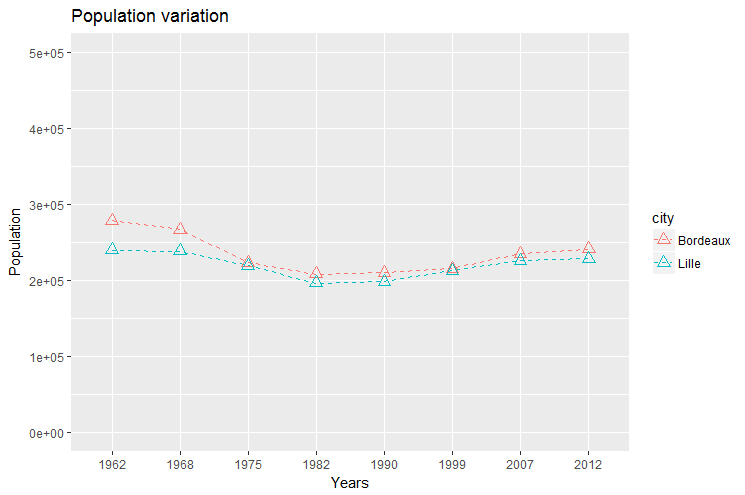
12.13.4. Ajuster les paramètres du graphique
Pour produire un graphique de qualité, l’affichage des données brutes n’est pas suffisant, même si ggplot2 construit par lui même une information pertinente, en produisant automatiquement une légende, des labels pour les axes, etc…
Il est nécessaire d’ajouter un titre, des labels autres que ceux produits par défaut à partir du nom des variables, une échelle adaptée, et plein d’autres choses encore.
Améliorer le graphique précédent :
en ajoutant un titre et des labels personnalisés
en fixant manuellement les limites de l’axe des abscisses
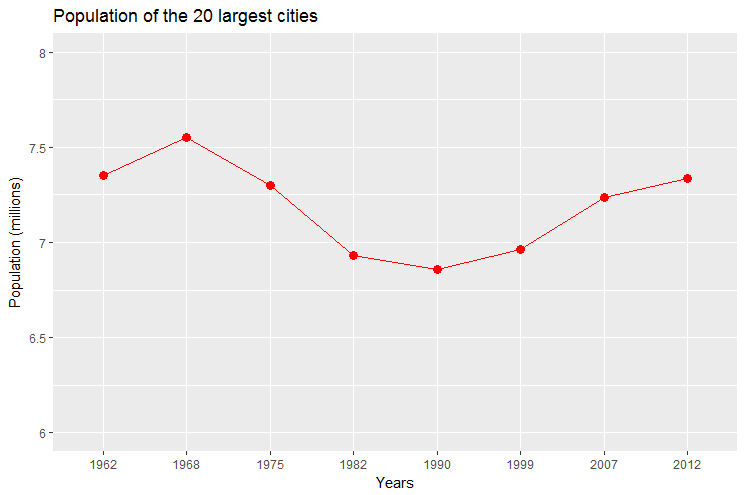
12.13.5. Réduction de données
ggplot2 utilise les données contenues dans la data.frame pour construire les graphiques, mais peut faire appel au layer stat pour effectuer une transformation statistique des données (on dit aussi réduction). Ce peut être le calcul d’une moyenne mean(), d’une somme sum(), etc…
Tracer l’évolution de la population dans l’ensemble des 20 plus grandes villes de France en utilisant la fonction stat_summary().
Le graphique doit ressembler à celui ci dessous.
12.13.6. Toutes les données
Essayons maintenant d’avoir une vue d’ensemble du jeu de données en affichant l’ensemble des données sur un seul et même graphique.
12.13.6.1. Courbes superposées
Quelles sont les instructions permettant de superposer les courbes et d’obtenir le graphique ci dessous ?
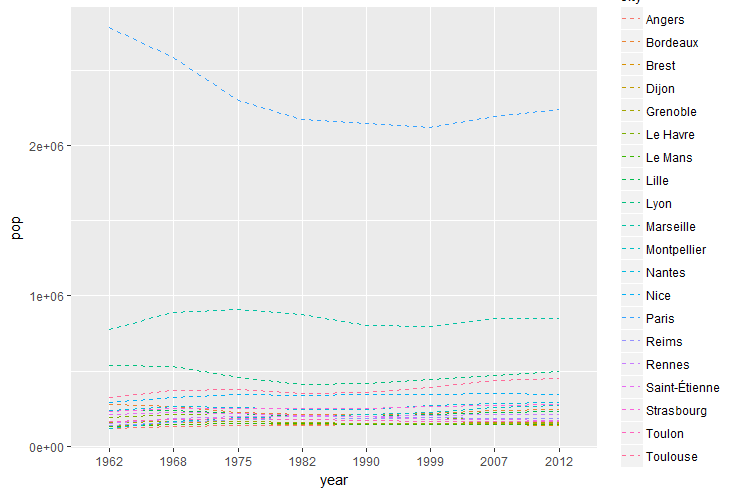
L’objectif est atteint mais le graphique est illisible.
12.13.6.2. Bar plot
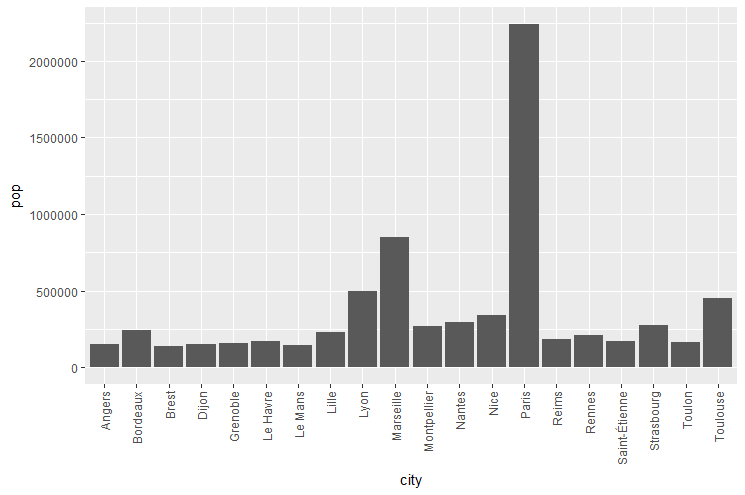
Construire un barplot présentant la population des 20 plus grandes villes de France pour l’année 1962.
Le résultat doit ressembler à ça:
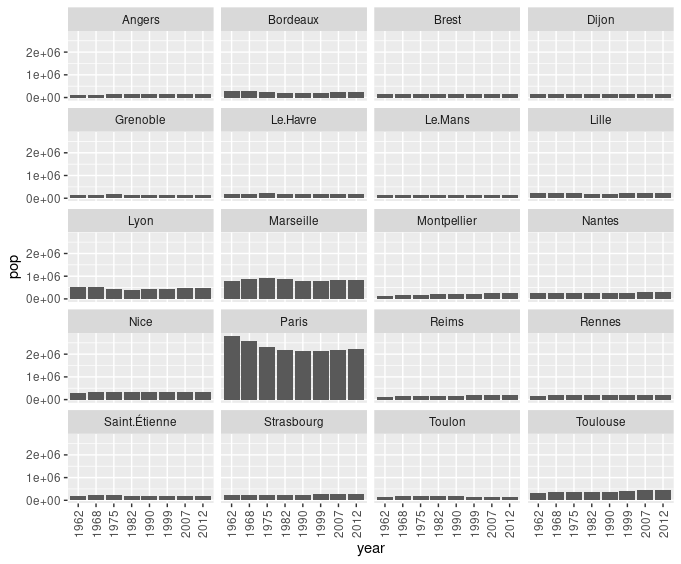
Utiliser le facetting pour présenter l’évolution de population pour chacune des villes.
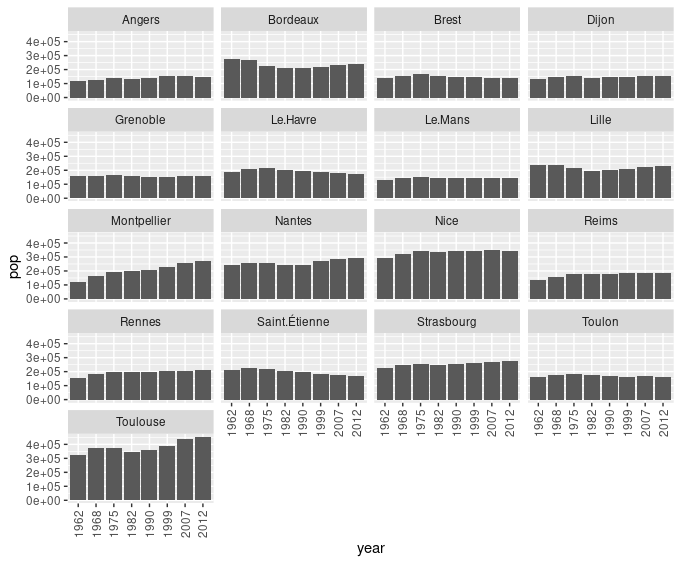
Exclure Paris, Marseille et Lyon pour présenter sur un même graphique uniquement des villes de taille à peu près comparables.
L’utilisation de la fonction bar_geom() produit une autre visualisation.
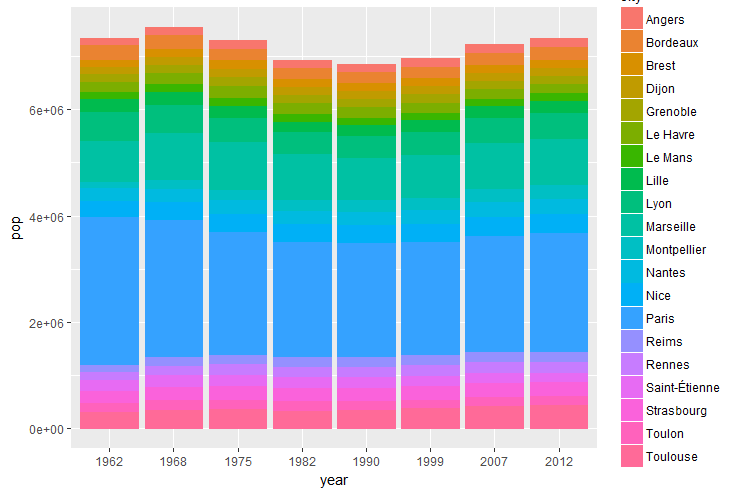
Ecrire les instructions permettant d’obtenir le graphique ci dessus.
Utiliser un grand nombre de paramètres sur un graphique n’est pas forcément judicieux (échelle de couleur difficile à lire, beaucoup de groupements, etc…). Le choix des villes comme paramètre n’est donc pas très pertinent.
Comme les données sont également paramétrées en fonction des années, on peut également utiliser la variable year.
Ecrire les instructions permettant de construire le graphique ci dessous.
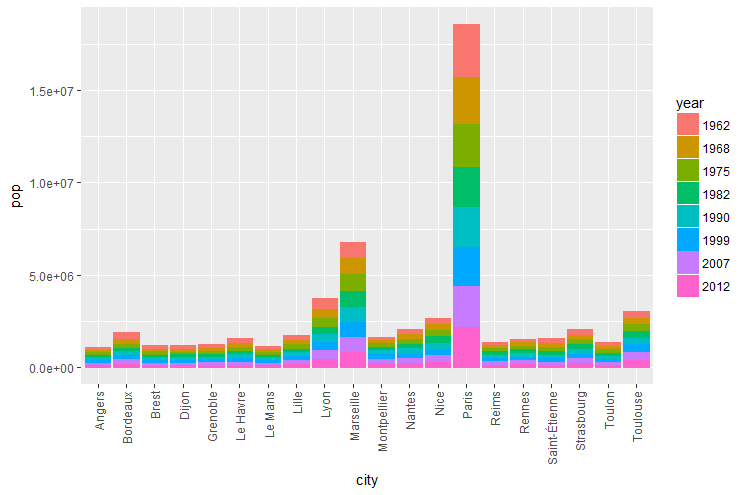
Ce graphique nous donne une information qualitative (les 4 villes les plus peuplées sont Paris, Marseille, Lyon et Toulouse, la population de Paris a diminué depuis les années 60, etc…). En fait le nombre important de données et de groupements (20 villes et 8 années) rend l’affichage complexe.
12.13.6.3. Heat map
Un autre choix peut être un graphique de type « heat map », pour lequel un gradient de couleur est utilisé. La fonction mise en jeu ici est geom_tile().
Ecrire les instructions pour construire le graphique ci dessous.
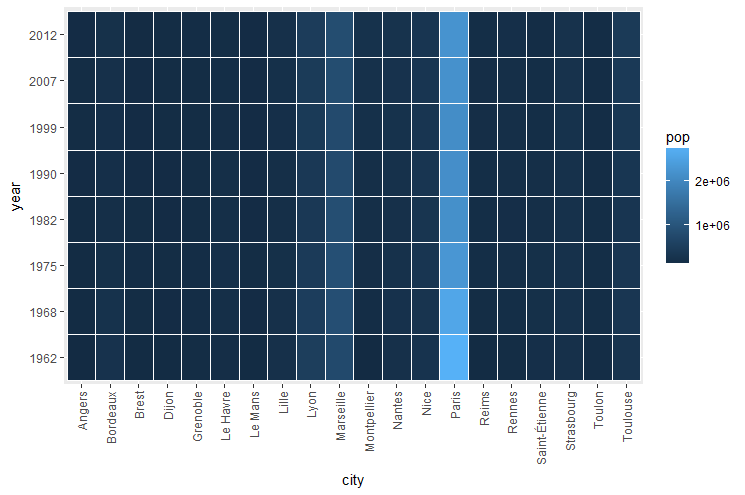
Ce type de graphique est cependant plus pertinent avec des données continues, ce qui n’est pas le cas ici. La représentation de la population de Paris nettement plus importante que celle des autres villes, est un réel challenge.
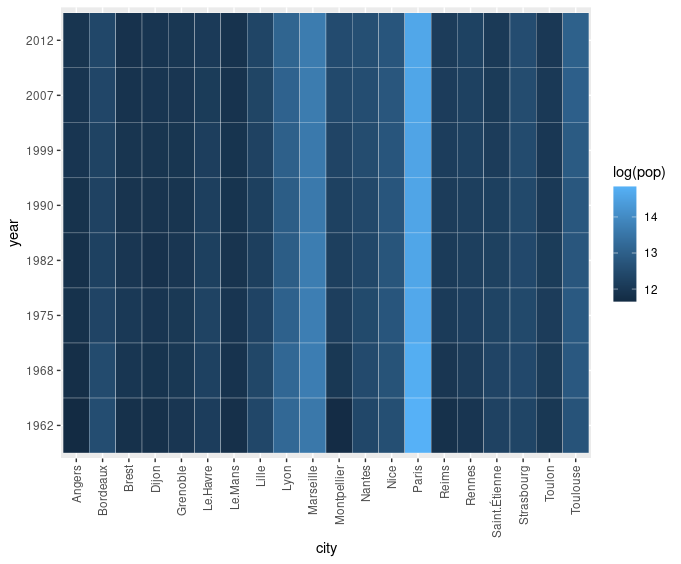
On pourrait penser à utiliser une fonction de compression de données, comme la fonction logarithmique par exemple, qui distribue le gradient de couleur entre des valeurs moins éloignées.
Ecrire les instructions affichant le log de la population.
Le graphique devrait ressembler à celui ci dessous.
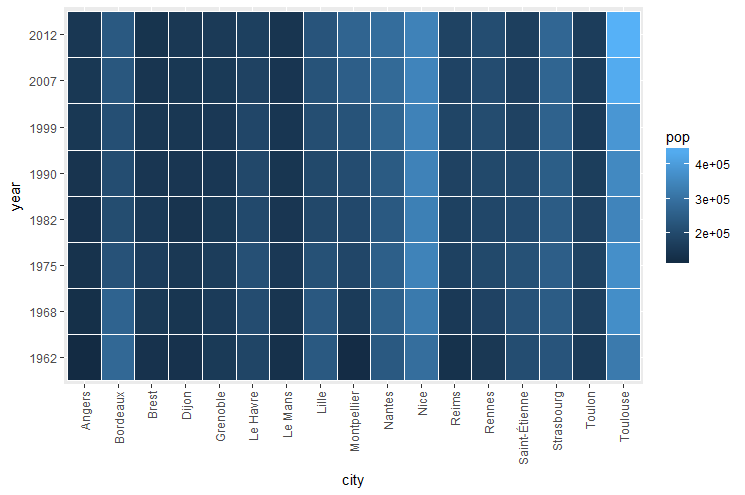
Une autre idée, déjà utilisée plus haut, est de retirer les données « déviantes ». Filtrer la data.frame passée à la fonction ggplot() pour obtenir le graphique ci dessous.
12.13.6.4. Dot plot
Un dot plot (ou scatter plot) peut être utilisé pour représenter une variable numérique.
Ecrire les instructions pour construire le graphique ci dessous.
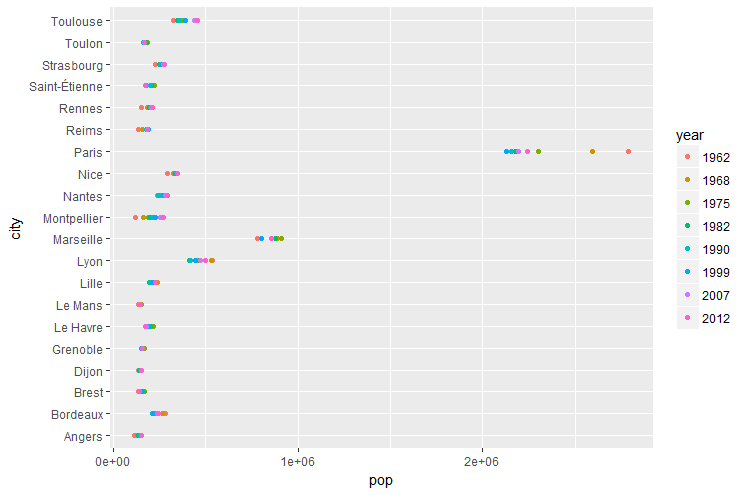
Ce graphique donne plusieurs informations:
le nombre d’habitants « moyen »
la dispersion (évolution) au fil des recensements
Quelles sont les villes dont le nombre d’habitants est resté relativement constant ? Ou au contraire ayant enregistré de grandes variations ? Quelles villes sont de taille comparable ?
On peut également imaginer « normaliser » les données pour comparer l’évolution de la population relative, indépendamment de la taille de la ville:
> p <- ggplot(dfl %>% group_by(city) %>% mutate(popn=pop/max(pop)), mapping=aes(y=city, x=popn, group=year, color=year, fill=year))
> p <- p + geom_point()
> p
Ecrire les instructions pour construire le graphique ci dessous. On utilisera les fonctions group_by() et mutate() (dans cet ordre là) pour grouper les données de la data.frame par ville avant de les normaliser. La fonction de normalisation utilisée sera:
x / max(x)
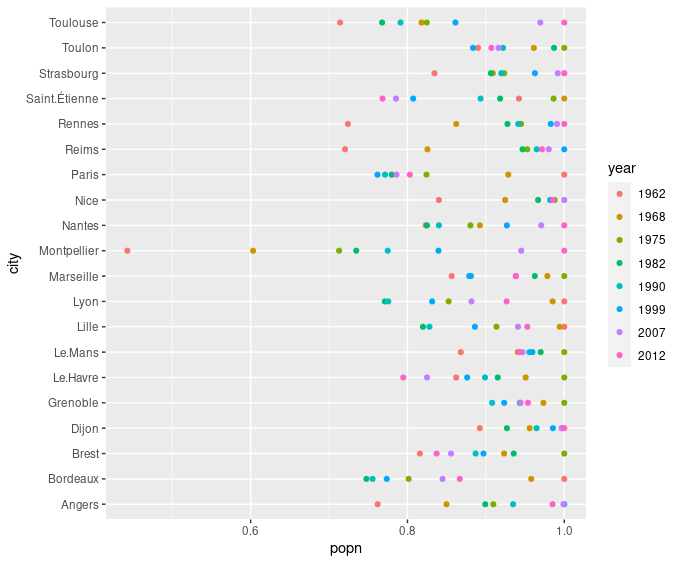
Quelle est la ville dont la population est resté la plus stable ? Quelle est la ville dont la population a le plus varié ?
12.14. Ressources additionnelles
Ce chapitre présente des exemples de flux de traitements des données basés sur les packages Tidyverse.
Il est fait une utilisation intensive des fonctions du package dplyr dont on consultera systématiquement la référence.
Au delà de la syntaxe des fonctions, les articles suivants recèlent une mine d’informations:
Grouped data aborde la problèmatique du groupement de données
Two-table verbs est utile pour combiner plusieurs data frames
dplyr <-> base R donne une équivalence entre les fonctions R core et leur équivalent dans l’éco système Tidyverse.
Column-wise operations donne des informations sur le traitement simultané de plusieurs variables
bien que R soit optimisé pour le traitement de variables, Row-wise operations fait le point sur la façon de traiter simultanément plusieurs observations.
Le Tidyverse design guide donne les bonnes pratiques de l’utilisation de l’éco système.
On trouvera également une information plus exhaustive pour les sujets suivants:
le temps et les dates : package lubridate. Une mise en oeuvre est disponible dans le chapitre Dates and times de l’excellent R for Data Science.
les chaines de caractères : package stringr. Une mise en oeuvre est disponible dans le chapitre Strings de l’excellent R for Data Science.